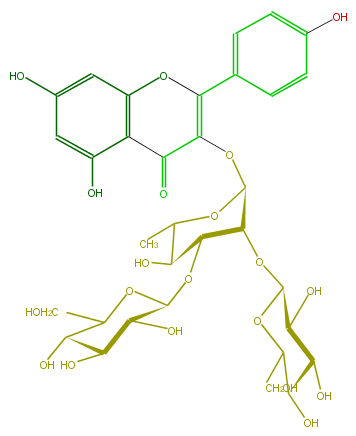
Mol:FL5FAAGS0085
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.1286 3.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1286 3.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1286 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1286 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8431 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8431 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5575 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5575 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5575 3.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5575 3.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8431 4.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8431 4.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4141 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4141 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 2.9918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 2.9918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0148 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0148 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0148 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0148 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 1.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 1.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4141 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4141 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4438 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4438 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4438 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4438 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 1.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 1.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 0.6235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 0.6235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1582 2.9918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1582 2.9918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0204 1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0204 1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1582 4.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1582 4.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 0.6422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 0.6422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0864 0.0292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0864 0.0292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1456 -0.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1456 -0.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4691 -0.2434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4691 -0.2434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2784 -0.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2784 -0.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3376 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3376 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7229 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7229 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6244 -0.7337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6244 -0.7337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2002 -1.0631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2002 -1.0631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6653 -0.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6653 -0.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7151 -0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7151 -0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2601 -2.2533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2601 -2.2533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5000 -2.5738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5000 -2.5738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9899 -1.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9899 -1.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2206 -1.6272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2206 -1.6272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9808 -1.3065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9808 -1.3065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4910 -1.9550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4910 -1.9550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6902 -2.7819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6902 -2.7819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1362 -2.7839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1362 -2.7839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1295 -2.1045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1295 -2.1045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1907 -3.3859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1907 -3.3859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6888 -2.7284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6888 -2.7284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1864 -2.0741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1864 -2.0741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0875 -1.2551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0875 -1.2551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5890 -1.9125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5890 -1.9125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0915 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0915 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5817 -3.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5817 -3.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8467 -3.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8467 -3.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6375 -1.3045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6375 -1.3045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3588 -1.7300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3588 -1.7300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9084 -1.7988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9084 -1.7988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7061 -3.2473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7061 -3.2473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5169 -4.2293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5169 -4.2293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 29 1 0 0 0 0 | + | 35 29 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 28 1 0 0 0 0 | + | 44 28 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 37 50 1 0 0 0 0 | + | 37 50 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 56 0.8677 -0.2250 | + | M SBV 1 56 0.8677 -0.2250 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 58 0.3853 0.6805 | + | M SBV 2 58 0.3853 0.6805 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0085 | + | ID FL5FAAGS0085 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES C(C(OC(C2OC(C6O)OC(CO)C(O)C6O)C(OC(C(=O)3)=C(c(c5)ccc(O)c5)Oc(c4)c(c(O)cc4O)3)OC(C2O)C)1)(O)C(O)C(C(CO)O1)O | + | SMILES C(C(OC(C2OC(C6O)OC(CO)C(O)C6O)C(OC(C(=O)3)=C(c(c5)ccc(O)c5)Oc(c4)c(c(O)cc4O)3)OC(C2O)C)1)(O)C(O)C(C(CO)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
1.1286 3.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1286 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8431 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5575 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5575 3.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8431 4.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4141 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 2.9918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0148 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0148 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 1.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4141 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 2.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4438 2.5793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4438 1.7543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 1.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 0.6235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1582 2.9918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0204 1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1582 4.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 0.6422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0864 0.0292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1456 -0.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4691 -0.2434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2784 -0.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3376 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7229 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6244 -0.7337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2002 -1.0631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6653 -0.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7151 -0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2601 -2.2533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5000 -2.5738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9899 -1.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2206 -1.6272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9808 -1.3065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4910 -1.9550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6902 -2.7819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1362 -2.7839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1295 -2.1045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1907 -3.3859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6888 -2.7284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1864 -2.0741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0875 -1.2551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5890 -1.9125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0915 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5817 -3.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8467 -3.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6375 -1.3045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3588 -1.7300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9084 -1.7988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7061 -3.2473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5169 -4.2293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
25 28 1 0 0 0 0
24 29 1 0 0 0 0
23 30 1 0 0 0 0
22 31 1 0 0 0 0
26 19 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 29 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 28 1 0 0 0 0
50 51 1 0 0 0 0
37 50 1 0 0 0 0
52 53 1 0 0 0 0
46 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 56
M SMT 1 ^ CH2OH
M SBV 1 56 0.8677 -0.2250
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 58
M SMT 2 ^ CH2OH
M SBV 2 58 0.3853 0.6805
S SKP 5
ID FL5FAAGS0085
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES C(C(OC(C2OC(C6O)OC(CO)C(O)C6O)C(OC(C(=O)3)=C(c(c5)ccc(O)c5)Oc(c4)c(c(O)cc4O)3)OC(C2O)C)1)(O)C(O)C(C(CO)O1)O
M END