Mol:FL5FAAGS0075
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.8489 1.5054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8489 1.5054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8489 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8489 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5634 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5634 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2779 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2779 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2779 1.5054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2779 1.5054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5634 1.9179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5634 1.9179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1344 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1344 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4200 0.6804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4200 0.6804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2945 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2945 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2945 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2945 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4200 -0.9696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4200 -0.9696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1344 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1344 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0090 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0090 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7234 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7234 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7234 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7234 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0090 -0.9696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0090 -0.9696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4200 -1.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4200 -1.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4365 0.4934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4365 0.4934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7408 -0.9072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7408 -0.9072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8786 1.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8786 1.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0090 -1.6692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0090 -1.6692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5970 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5970 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3275 -0.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3275 -0.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1508 0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1508 0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4094 0.7851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4094 0.7851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6788 0.4018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6788 0.4018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8554 -0.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8554 -0.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3492 -0.7669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3492 -0.7669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3007 -1.4001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3007 -1.4001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8141 -1.9179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8141 -1.9179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1965 -1.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1965 -1.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8051 0.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8051 0.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9428 -0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9428 -0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3553 -0.8950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3553 -0.8950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5620 -0.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5620 -0.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7640 -0.8774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7640 -0.8774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3514 -0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3514 -0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1447 -0.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1447 -0.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7068 -1.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7068 -1.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9956 -1.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9956 -1.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8051 -0.7802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8051 -0.7802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6565 -0.2633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6565 -0.2633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 18 1 0 0 0 0 | + | 37 18 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
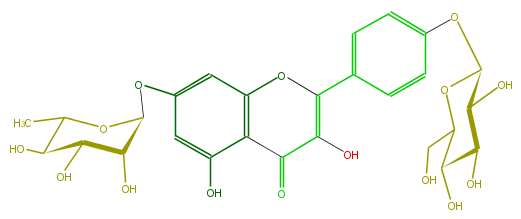
| − | ID FL5FAAGS0075 | + | ID FL5FAAGS0075 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c4)(ccc(OC(O5)C(O)C(O)C(O)C5CO)c4)C(=C(O)3)Oc(c2C3=O)cc(cc2O)OC(C(O)1)OC(C)C(O)C1O | + | SMILES c(c4)(ccc(OC(O5)C(O)C(O)C(O)C5CO)c4)C(=C(O)3)Oc(c2C3=O)cc(cc2O)OC(C(O)1)OC(C)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
1.8489 1.5054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8489 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5634 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2779 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2779 1.5054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5634 1.9179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1344 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4200 0.6804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2945 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2945 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4200 -0.9696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1344 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0090 0.6804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7234 0.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7234 -0.5571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0090 -0.9696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4200 -1.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4365 0.4934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7408 -0.9072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8786 1.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0090 -1.6692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5970 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3275 -0.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1508 0.0017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4094 0.7851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6788 0.4018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8554 -0.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3492 -0.7669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3007 -1.4001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8141 -1.9179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1965 -1.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8051 0.5045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9428 -0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3553 -0.8950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5620 -0.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7640 -0.8774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3514 -0.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1447 -0.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7068 -1.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9956 -1.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8051 -0.7802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6565 -0.2633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 20 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 18 1 0 0 0 0
S SKP 5
ID FL5FAAGS0075
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c4)(ccc(OC(O5)C(O)C(O)C(O)C5CO)c4)C(=C(O)3)Oc(c2C3=O)cc(cc2O)OC(C(O)1)OC(C)C(O)C1O
M END