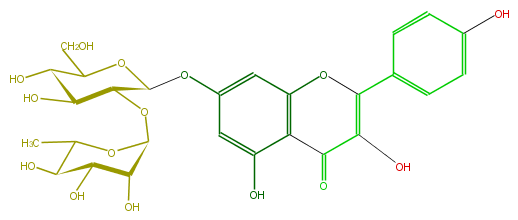
Mol:FL5FAAGS0035
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6036 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6036 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6036 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6036 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1109 -0.9224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1109 -0.9224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8253 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8253 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8253 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8253 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1109 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1109 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5398 -0.9224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5398 -0.9224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2542 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2542 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2542 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2542 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5398 0.7277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5398 0.7277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5398 -1.5656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5398 -1.5656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9684 0.7275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9684 0.7275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6966 0.3070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6966 0.3070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4249 0.7275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4249 0.7275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4249 1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4249 1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6966 1.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6966 1.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9684 1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9684 1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1109 -1.7471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1109 -1.7471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3178 0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3178 0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1528 1.9886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1528 1.9886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1131 -1.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1131 -1.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0933 0.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0933 0.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5725 0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5725 0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8227 0.4289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8227 0.4289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0412 0.4203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0412 0.4203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6249 0.9626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6249 0.9626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3909 0.6875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3909 0.6875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6995 0.6344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6995 0.6344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4035 0.2408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4035 0.2408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1442 -0.0461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1442 -0.0461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5764 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5764 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9679 -1.3473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9679 -1.3473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2150 -1.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2150 -1.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4577 -1.3473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4577 -1.3473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0662 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0662 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8191 -0.8845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8191 -0.8845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3847 -0.6857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3847 -0.6857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4750 -1.1612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4750 -1.1612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6063 -1.7676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6063 -1.7676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4769 -1.9887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4769 -1.9887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8997 1.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8997 1.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1528 1.6062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1528 1.6062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.5088 -0.6373 | + | M SBV 1 46 0.5088 -0.6373 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0035 | + | ID FL5FAAGS0035 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES Oc(c5)ccc(c5)C(=C(O)4)Oc(c1C4=O)cc(OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)cc1O | + | SMILES Oc(c5)ccc(c5)C(=C(O)4)Oc(c1C4=O)cc(OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.6036 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6036 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1109 -0.9224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8253 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8253 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1109 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5398 -0.9224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2542 -0.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2542 0.3151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5398 0.7277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5398 -1.5656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9684 0.7275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6966 0.3070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4249 0.7275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4249 1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6966 1.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9684 1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1109 -1.7471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3178 0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1528 1.9886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1131 -1.1636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0933 0.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5725 0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8227 0.4289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0412 0.4203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6249 0.9626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3909 0.6875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6995 0.6344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4035 0.2408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1442 -0.0461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5764 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9679 -1.3473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2150 -1.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4577 -1.3473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0662 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8191 -0.8845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3847 -0.6857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4750 -1.1612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6063 -1.7676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4769 -1.9887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8997 1.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1528 1.6062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 30 1 0 0 0 0
25 19 1 0 0 0 0
41 42 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.5088 -0.6373
S SKP 5
ID FL5FAAGS0035
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES Oc(c5)ccc(c5)C(=C(O)4)Oc(c1C4=O)cc(OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)cc1O
M END