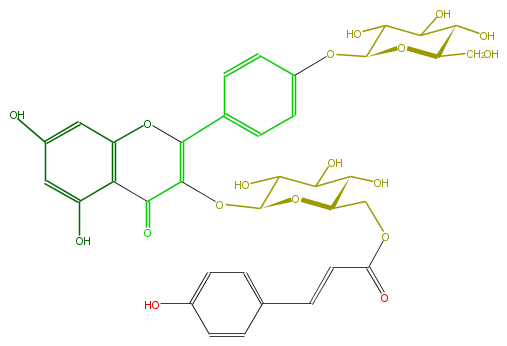
Mol:FL5FAAGL0091
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.5844 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5844 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5844 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5844 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8696 -0.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8696 -0.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1549 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1549 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1549 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1549 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8696 1.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8696 1.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4401 -0.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4401 -0.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7253 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7253 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7253 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7253 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4401 1.0710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4401 1.0710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4401 -1.2232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4401 -1.2232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8253 1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8253 1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0968 0.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0968 0.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6316 1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6316 1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6316 2.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6316 2.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0968 2.4915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0968 2.4915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8253 2.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8253 2.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8696 -1.4047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8696 -1.4047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4112 2.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4112 2.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2468 1.2413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2468 1.2413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9167 -0.6340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9167 -0.6340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3227 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3227 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0637 -0.7233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0637 -0.7233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6795 -0.5108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6795 -0.5108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4273 -0.7233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4273 -0.7233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8138 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8138 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0705 -0.2662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0705 -0.2662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3781 -0.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3781 -0.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4192 0.2418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4192 0.2418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3936 -0.1710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3936 -0.1710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1387 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1387 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5427 3.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5427 3.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1561 2.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1561 2.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8994 2.6661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8994 2.6661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6472 2.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6472 2.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0338 3.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0338 3.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2905 2.9108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2905 2.9108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9669 2.9462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9669 2.9462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7023 3.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7023 3.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6134 2.9029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6134 2.9029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5680 -1.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5680 -1.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1855 -1.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1855 -1.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4296 -1.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4296 -1.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9888 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9888 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0291 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0291 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4036 -2.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4036 -2.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1524 -2.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1524 -2.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5270 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5270 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1524 -3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1524 -3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4036 -3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4036 -3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2742 -2.7276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2742 -2.7276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5493 -2.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5493 -2.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3290 2.5279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3290 2.5279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2468 1.9979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2468 1.9979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 33 19 1 0 0 0 0 | + | 33 19 1 0 0 0 0 |
| − | 31 41 1 0 0 0 0 | + | 31 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 42 52 2 0 0 0 0 | + | 42 52 2 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 35 53 1 0 0 0 0 | + | 35 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 59 -0.6818 -0.0742 | + | M SBV 1 59 -0.6818 -0.0742 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0091 | + | ID FL5FAAGL0091 |
| − | FORMULA C36H36O18 | + | FORMULA C36H36O18 |
| − | EXACTMASS 756.190164348 | + | EXACTMASS 756.190164348 |
| − | AVERAGEMASS 756.6602399999999 | + | AVERAGEMASS 756.6602399999999 |
| − | SMILES Oc(c6)c(C(=O)3)c(cc6O)OC(=C3OC(O4)C(C(C(O)C4COC(=O)C=Cc(c5)ccc(c5)O)O)O)c(c1)ccc(OC(C(O)2)OC(CO)C(O)C2O)c1 | + | SMILES Oc(c6)c(C(=O)3)c(cc6O)OC(=C3OC(O4)C(C(C(O)C4COC(=O)C=Cc(c5)ccc(c5)O)O)O)c(c1)ccc(OC(C(O)2)OC(CO)C(O)C2O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-4.5844 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5844 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8696 -0.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1549 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1549 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8696 1.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4401 -0.5797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7253 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7253 0.6583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4401 1.0710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4401 -1.2232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8253 1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0968 0.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6316 1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6316 2.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0968 2.4915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8253 2.0710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8696 -1.4047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4112 2.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2468 1.2413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9167 -0.6340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3227 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0637 -0.7233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6795 -0.5108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4273 -0.7233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8138 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0705 -0.2662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3781 -0.2295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4192 0.2418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3936 -0.1710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1387 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5427 3.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1561 2.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8994 2.6661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6472 2.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0338 3.1231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2905 2.9108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9669 2.9462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7023 3.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6134 2.9029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5680 -1.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1855 -1.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4296 -1.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9888 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0291 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4036 -2.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1524 -2.0792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5270 -2.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1524 -3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4036 -3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2742 -2.7276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5493 -2.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3290 2.5279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2468 1.9979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 21 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
33 19 1 0 0 0 0
31 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
42 52 2 0 0 0 0
53 54 1 0 0 0 0
35 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 59
M SMT 1 CH2OH
M SBV 1 59 -0.6818 -0.0742
S SKP 5
ID FL5FAAGL0091
FORMULA C36H36O18
EXACTMASS 756.190164348
AVERAGEMASS 756.6602399999999
SMILES Oc(c6)c(C(=O)3)c(cc6O)OC(=C3OC(O4)C(C(C(O)C4COC(=O)C=Cc(c5)ccc(c5)O)O)O)c(c1)ccc(OC(C(O)2)OC(CO)C(O)C2O)c1
M END