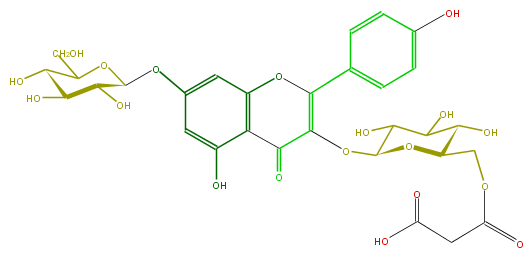
Mol:FL5FAAGL0084
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9329 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9329 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9329 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9329 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2185 -0.4713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2185 -0.4713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5040 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5040 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5040 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5040 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2185 1.1787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2185 1.1787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2104 -0.4713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2104 -0.4713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9249 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9249 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9249 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9249 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2104 1.1787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2104 1.1787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2104 -1.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2104 -1.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8246 1.3374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8246 1.3374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5526 0.9171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5526 0.9171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2808 1.3374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2808 1.3374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2808 2.1783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2808 2.1783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5526 2.5986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5526 2.5986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8246 2.1783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8246 2.1783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2185 -1.2960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2185 -1.2960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0602 2.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0602 2.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5951 1.3489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5951 1.3489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7332 -0.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7332 -0.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8007 0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8007 0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4144 -0.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4144 -0.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1574 -0.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1574 -0.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9049 -0.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9049 -0.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2913 0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2913 0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5482 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5482 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1877 -0.0892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1877 -0.0892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9394 0.3161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9394 0.3161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9454 -0.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9454 -0.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6454 -0.5259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6454 -0.5259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8883 -1.3205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8883 -1.3205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8883 -2.1682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8883 -2.1682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6850 -2.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6850 -2.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1350 -2.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1350 -2.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1090 1.3160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1090 1.3160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6323 0.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6323 0.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9458 0.9537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9458 0.9537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2835 0.9608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2835 0.9608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7648 1.4423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7648 1.4423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3654 1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3654 1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6850 1.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6850 1.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2916 0.6904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2916 0.6904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4068 0.5188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4068 0.5188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3450 -2.1471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3450 -2.1471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6193 -2.5660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6193 -2.5660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3450 -1.5046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3450 -1.5046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8757 1.7186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8757 1.7186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8522 1.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8522 1.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 39 20 1 0 0 0 0 | + | 39 20 1 0 0 0 0 |
| − | 35 45 1 0 0 0 0 | + | 35 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 45 47 2 0 0 0 0 | + | 45 47 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 41 48 1 0 0 0 0 | + | 41 48 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 48 49 | + | M SAL 1 2 48 49 |
| − | M SBL 1 1 53 | + | M SBL 1 1 53 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 53 0.5103 -0.5933 | + | M SBV 1 53 0.5103 -0.5933 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0084 | + | ID FL5FAAGL0084 |
| − | FORMULA C30H32O19 | + | FORMULA C30H32O19 |
| − | EXACTMASS 696.153778842 | + | EXACTMASS 696.153778842 |
| − | AVERAGEMASS 696.56368 | + | AVERAGEMASS 696.56368 |
| − | SMILES c(c5)(cc(c(c5O)2)OC(c(c4)ccc(c4)O)=C(OC(O3)C(C(C(O)C3COC(CC(O)=O)=O)O)O)C(=O)2)OC(O1)C(O)C(O)C(O)C1CO | + | SMILES c(c5)(cc(c(c5O)2)OC(c(c4)ccc(c4)O)=C(OC(O3)C(C(C(O)C3COC(CC(O)=O)=O)O)O)C(=O)2)OC(O1)C(O)C(O)C(O)C1CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-1.9329 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9329 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2185 -0.4713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5040 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5040 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2185 1.1787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2104 -0.4713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9249 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9249 0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2104 1.1787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2104 -1.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8246 1.3374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5526 0.9171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2808 1.3374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2808 2.1783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5526 2.5986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8246 2.1783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2185 -1.2960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0602 2.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5951 1.3489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7332 -0.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8007 0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4144 -0.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1574 -0.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9049 -0.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2913 0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5482 -0.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1877 -0.0892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9394 0.3161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9454 -0.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6454 -0.5259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8883 -1.3205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8883 -2.1682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6850 -2.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1350 -2.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1090 1.3160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6323 0.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9458 0.9537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2835 0.9608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7648 1.4423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3654 1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6850 1.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2916 0.6904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4068 0.5188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3450 -2.1471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6193 -2.5660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3450 -1.5046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8757 1.7186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8522 1.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 21 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
39 20 1 0 0 0 0
35 45 1 0 0 0 0
45 46 1 0 0 0 0
45 47 2 0 0 0 0
48 49 1 0 0 0 0
41 48 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 48 49
M SBL 1 1 53
M SMT 1 ^CH2OH
M SBV 1 53 0.5103 -0.5933
S SKP 5
ID FL5FAAGL0084
FORMULA C30H32O19
EXACTMASS 696.153778842
AVERAGEMASS 696.56368
SMILES c(c5)(cc(c(c5O)2)OC(c(c4)ccc(c4)O)=C(OC(O3)C(C(C(O)C3COC(CC(O)=O)=O)O)O)C(=O)2)OC(O1)C(O)C(O)C(O)C1CO
M END