Mol:FL5FAAGL0028
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1129 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1129 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1129 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1129 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5566 1.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5566 1.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0003 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0003 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0003 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0003 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5566 2.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5566 2.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4440 1.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4440 1.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8877 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8877 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8877 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8877 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4440 2.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4440 2.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4440 0.5249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4440 0.5249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3316 2.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3316 2.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2354 1.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2354 1.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8023 2.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8023 2.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8023 2.9650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8023 2.9650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2354 3.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2354 3.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3316 2.9650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3316 2.9650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5566 0.3836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5566 0.3836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6690 2.3103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6690 2.3103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3691 3.2922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3691 3.2922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4935 0.9580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4935 0.9580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9095 -0.6908 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.9095 -0.6908 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.6349 -0.6908 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.6349 -0.6908 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.1136 -0.1865 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.1136 -0.1865 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.9353 0.5209 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9353 0.5209 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2099 0.5210 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2099 0.5210 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.7312 0.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7312 0.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6470 -0.1865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6470 -0.1865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8019 -1.3141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8019 -1.3141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4862 0.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4862 0.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2171 -1.8386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2171 -1.8386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4263 1.5136 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.4263 1.5136 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.8687 0.9297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.8687 0.9297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5058 1.1774 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.5058 1.1774 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1205 1.1840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1205 1.1840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6738 1.6308 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.6738 1.6308 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1165 1.3367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1165 1.3367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1791 0.8961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1791 0.8961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8708 0.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8708 0.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6690 1.5007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6690 1.5007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8653 -2.3402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8653 -2.3402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5218 -2.4226 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.5218 -2.4226 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.1164 -2.9578 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1164 -2.9578 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5325 -2.7308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5325 -2.7308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.0760 -2.7375 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.0760 -2.7375 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.3785 -2.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3785 -2.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9749 -2.5294 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.9749 -2.5294 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.0431 -2.3770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0431 -2.3770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4991 -3.4616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4991 -3.4616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1980 -3.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1980 -3.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4307 2.2843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4307 2.2843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2433 3.2666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2433 3.2666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1955 -1.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1955 -1.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1712 -1.6043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1712 -1.6043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 41 31 1 0 0 0 0 | + | 41 31 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 41 1 0 0 0 0 | + | 45 41 1 0 0 0 0 |
| − | 36 51 1 0 0 0 0 | + | 36 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 58 -1.1955 -1.8233 | + | M SVB 2 58 -1.1955 -1.8233 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 56 3.2572 1.3054 | + | M SVB 1 56 3.2572 1.3054 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0028 | + | ID FL5FAAGL0028 |
| − | KNApSAcK_ID C00005215 | + | KNApSAcK_ID C00005215 |
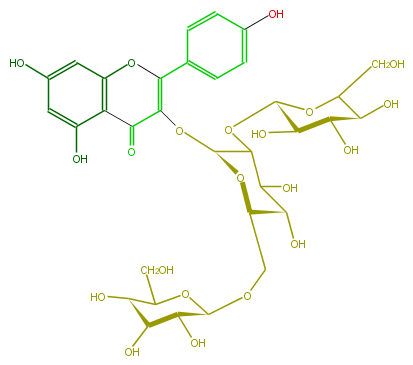
| − | NAME Kaempferol 3-glucosyl-(1->2)-gentiobioside | + | NAME Kaempferol 3-glucosyl-(1->2)-gentiobioside |
| − | CAS_RN 55696-59-8 | + | CAS_RN 55696-59-8 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES O=C(C(O[C@H](O4)C(O[C@H]([C@@H]6O)OC(CO)[C@H]([C@@H](O)6)O)C([C@H]([C@H]4CO[C@H](O5)[C@@H](O)[C@H]([C@@H](O)C5CO)O)O)O)=1)c(c(O)3)c(cc(c3)O)OC(c(c2)ccc(O)c2)1 | + | SMILES O=C(C(O[C@H](O4)C(O[C@H]([C@@H]6O)OC(CO)[C@H]([C@@H](O)6)O)C([C@H]([C@H]4CO[C@H](O5)[C@@H](O)[C@H]([C@@H](O)C5CO)O)O)O)=1)c(c(O)3)c(cc(c3)O)OC(c(c2)ccc(O)c2)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-3.1129 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1129 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5566 1.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0003 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0003 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5566 2.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4440 1.0257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8877 1.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8877 1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4440 2.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4440 0.5249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3316 2.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2354 1.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8023 2.3103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8023 2.9650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2354 3.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3316 2.9650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5566 0.3836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6690 2.3103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3691 3.2922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4935 0.9580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9095 -0.6908 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.6349 -0.6908 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.1136 -0.1865 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.9353 0.5209 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2099 0.5210 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.7312 0.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6470 -0.1865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8019 -1.3141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4862 0.9700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2171 -1.8386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4263 1.5136 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.8687 0.9297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5058 1.1774 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1205 1.1840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6738 1.6308 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1165 1.3367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1791 0.8961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8708 0.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6690 1.5007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8653 -2.3402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5218 -2.4226 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.1164 -2.9578 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5325 -2.7308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.0760 -2.7375 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.3785 -2.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9749 -2.5294 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.0431 -2.3770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4991 -3.4616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1980 -3.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4307 2.2843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2433 3.2666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1955 -1.8233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1712 -1.6043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
26 21 1 0 0 0 0
25 30 1 0 0 0 0
22 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
30 32 1 0 0 0 0
35 40 1 0 0 0 0
41 31 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 41 1 0 0 0 0
36 51 1 0 0 0 0
51 52 1 0 0 0 0
47 53 1 0 0 0 0
53 54 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 58
M SMT 2 CH2OH
M SVB 2 58 -1.1955 -1.8233
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 56
M SMT 1 CH2OH
M SVB 1 56 3.2572 1.3054
S SKP 8
ID FL5FAAGL0028
KNApSAcK_ID C00005215
NAME Kaempferol 3-glucosyl-(1->2)-gentiobioside
CAS_RN 55696-59-8
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES O=C(C(O[C@H](O4)C(O[C@H]([C@@H]6O)OC(CO)[C@H]([C@@H](O)6)O)C([C@H]([C@H]4CO[C@H](O5)[C@@H](O)[C@H]([C@@H](O)C5CO)O)O)O)=1)c(c(O)3)c(cc(c3)O)OC(c(c2)ccc(O)c2)1
M END