Mol:FL5FAAGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1751 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1751 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1751 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1751 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4607 0.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4607 0.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7463 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7463 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7463 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7463 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4607 2.5123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4607 2.5123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0319 0.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0319 0.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3174 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3174 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3174 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3174 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0319 2.5123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0319 2.5123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0319 0.1288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0319 0.1288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3967 2.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3967 2.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1249 2.0918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1249 2.0918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8531 2.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8531 2.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8531 3.3529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8531 3.3529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1249 3.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1249 3.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3967 3.3529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3967 3.3529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4607 0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4607 0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8893 2.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8893 2.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5810 3.7733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5810 3.7733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2069 -1.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2069 -1.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0137 -2.2838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0137 -2.2838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7577 -1.3880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7577 -1.3880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0137 -0.4867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0137 -0.4867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2069 -0.0208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2069 -0.0208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4628 -0.9167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4628 -0.9167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5551 0.8105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5551 0.8105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9470 0.4599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9470 0.4599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3013 -1.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3013 -1.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0137 -2.7188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0137 -2.7188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4176 1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4176 1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9782 0.4805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9782 0.4805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9682 0.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9682 0.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5251 1.1199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5251 1.1199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4732 1.5736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4732 1.5736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0012 1.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0012 1.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9682 1.0374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9682 1.0374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9682 -0.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9682 -0.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8893 -0.4909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8893 -0.4909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5301 -3.0242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5301 -3.0242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2793 -3.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2793 -3.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 27 8 1 0 0 0 0 | + | 27 8 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 21 40 1 0 0 0 0 | + | 21 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 43 0.0000 0.7040 | + | M SBV 1 43 0.0000 0.7040 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 45 -0.3232 1.2062 | + | M SBV 2 45 -0.3232 1.2062 |
| − | S SKP 5 | + | S SKP 5 |
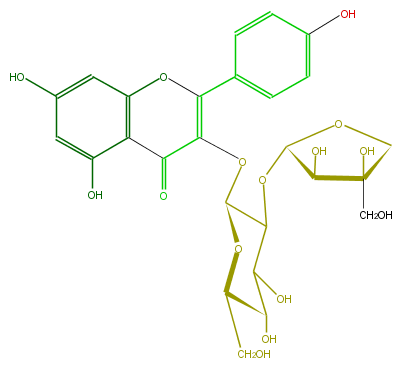
| − | ID FL5FAAGL0003 | + | ID FL5FAAGL0003 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES c(O2)(c(C(C(OC(O5)C(C(O)C(C(CO)5)O)OC(C(O)4)OCC4(O)CO)=C2c(c3)ccc(c3)O)=O)1)cc(O)cc1O | + | SMILES c(O2)(c(C(C(OC(O5)C(C(O)C(C(CO)5)O)OC(C(O)4)OCC4(O)CO)=C2c(c3)ccc(c3)O)=O)1)cc(O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-3.1751 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1751 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4607 0.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7463 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7463 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4607 2.5123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0319 0.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3174 1.2749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3174 2.0999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0319 2.5123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0319 0.1288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3967 2.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1249 2.0918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8531 2.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8531 3.3529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1249 3.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3967 3.3529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4607 0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8893 2.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5810 3.7733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2069 -1.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0137 -2.2838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7577 -1.3880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0137 -0.4867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2069 -0.0208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4628 -0.9167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5551 0.8105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9470 0.4599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3013 -1.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0137 -2.7188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4176 1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9782 0.4805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9682 0.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5251 1.1199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4732 1.5736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0012 1.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9682 1.0374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9682 -0.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8893 -0.4909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5301 -3.0242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2793 -3.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
22 30 1 0 0 0 0
27 8 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 31 1 0 0 0 0
32 36 1 0 0 0 0
28 31 1 0 0 0 0
33 37 1 0 0 0 0
38 39 1 0 0 0 0
33 38 1 0 0 0 0
40 41 1 0 0 0 0
21 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 CH2OH
M SBV 1 43 0.0000 0.7040
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 CH2OH
M SBV 2 45 -0.3232 1.2062
S SKP 5
ID FL5FAAGL0003
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES c(O2)(c(C(C(OC(O5)C(C(O)C(C(CO)5)O)OC(C(O)4)OCC4(O)CO)=C2c(c3)ccc(c3)O)=O)1)cc(O)cc1O
M END