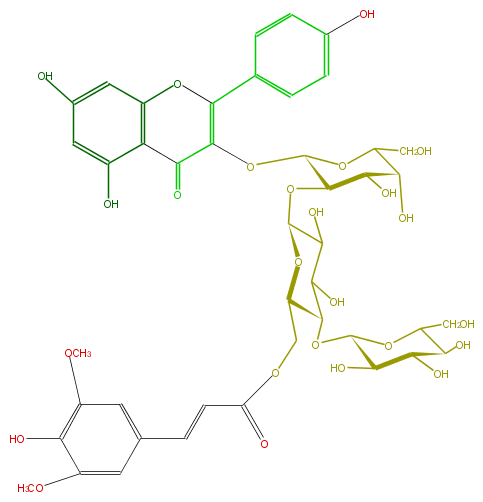
Mol:FL5FAAGA0037
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 69 75 0 0 0 0 0 0 0 0999 V2000 | + | 69 75 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.7607 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7607 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7607 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7607 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0461 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0461 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3316 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3316 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3316 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3316 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0461 3.7076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0461 3.7076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6171 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6171 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9026 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9026 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9026 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9026 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6171 3.7076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6171 3.7076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6171 1.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6171 1.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0030 3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0030 3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7253 3.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7253 3.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4535 3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4535 3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4535 4.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4535 4.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7253 5.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7253 5.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0030 4.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0030 4.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0461 1.2328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0461 1.2328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2329 5.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2329 5.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4230 3.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4230 3.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0942 2.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0942 2.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9905 2.2168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9905 2.2168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5064 1.5357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5064 1.5357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2495 1.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2495 1.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9664 1.8324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9664 1.8324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4455 2.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4455 2.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7954 2.0104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7954 2.0104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7282 1.5342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7282 1.5342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6765 1.4593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6765 1.4593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0233 0.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0233 0.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3772 0.4081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3772 0.4081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6665 0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6665 0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8921 0.0293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8921 0.0293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6665 -0.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6665 -0.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3772 -1.1747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3772 -1.1747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1518 -0.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1518 -0.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1744 1.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1744 1.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6126 -0.7945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6126 -0.7945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2432 -1.6747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2432 -1.6747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8324 -1.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8324 -1.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4149 -2.2560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4149 -2.2560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2699 -2.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2699 -2.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1778 -3.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1778 -3.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0638 -2.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0638 -2.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4572 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4572 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3889 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3889 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7992 -2.9121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7992 -2.9121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6194 -2.9121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6194 -2.9121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0295 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0295 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6194 -4.3328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6194 -4.3328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7992 -4.3328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7992 -4.3328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8490 -3.6224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8490 -3.6224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9406 -1.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9406 -1.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4565 -2.1730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4565 -2.1730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1995 -1.8840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1995 -1.8840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9165 -1.8763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9165 -1.8763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3955 -1.3552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3955 -1.3552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7455 -1.6983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7455 -1.6983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7787 -2.1531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7787 -2.1531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7503 -2.2700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7503 -2.2700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2165 -1.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2165 -1.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9313 -1.2421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9313 -1.2421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8490 -1.7719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8490 -1.7719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8682 -1.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8682 -1.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9505 -2.3632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9505 -2.3632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0403 2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0403 2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9580 1.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9580 1.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4581 -4.6275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4581 -4.6275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5404 -5.1573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5404 -5.1573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 32 28 1 0 0 0 0 | + | 32 28 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 49 52 1 0 0 0 0 | + | 49 52 1 0 0 0 0 |
| − | 53 54 1 1 0 0 0 | + | 53 54 1 1 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 56 55 1 1 0 0 0 | + | 56 55 1 1 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 54 59 1 0 0 0 0 | + | 54 59 1 0 0 0 0 |
| − | 55 60 1 0 0 0 0 | + | 55 60 1 0 0 0 0 |
| − | 56 61 1 0 0 0 0 | + | 56 61 1 0 0 0 0 |
| − | 39 53 1 0 0 0 0 | + | 39 53 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 57 62 1 0 0 0 0 | + | 57 62 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 48 64 1 0 0 0 0 | + | 48 64 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 26 66 1 0 0 0 0 | + | 26 66 1 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | 50 68 1 0 0 0 0 | + | 50 68 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 69 | + | M SBL 1 1 69 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 69 -0.5357 -0.1131 | + | M SBV 1 69 -0.5357 -0.1131 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 64 65 | + | M SAL 2 2 64 65 |
| − | M SBL 2 1 71 | + | M SBL 2 1 71 |
| − | M SMT 2 ^OCH3 | + | M SMT 2 ^OCH3 |
| − | M SBV 2 71 0.2488 -1.0786 | + | M SBV 2 71 0.2488 -1.0786 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 66 67 | + | M SAL 3 2 66 67 |
| − | M SBL 3 1 73 | + | M SBL 3 1 73 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 73 -0.5949 0.0285 | + | M SBV 3 73 -0.5949 0.0285 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 68 69 | + | M SAL 4 2 68 69 |
| − | M SBL 4 1 75 | + | M SBL 4 1 75 |
| − | M SMT 4 ^OCH3 | + | M SMT 4 ^OCH3 |
| − | M SBV 4 75 0.8387 0.2948 | + | M SBV 4 75 0.8387 0.2948 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGA0037 | + | ID FL5FAAGA0037 |
| − | FORMULA C44H50O25 | + | FORMULA C44H50O25 |
| − | EXACTMASS 978.26411715 | + | EXACTMASS 978.26411715 |
| − | AVERAGEMASS 978.8528000000001 | + | AVERAGEMASS 978.8528000000001 |
| − | SMILES C(C(O1)C(O)C(C(OC(O6)C(O)C(O)C(C(COC(=O)C=Cc(c7)cc(c(O)c7OC)OC)6)OC(O5)C(C(O)C(O)C5CO)O)C1OC(=C2c(c4)ccc(c4)O)C(c(c3O)c(cc(c3)O)O2)=O)O)O | + | SMILES C(C(O1)C(O)C(C(OC(O6)C(O)C(O)C(C(COC(=O)C=Cc(c7)cc(c(O)c7OC)OC)6)OC(O5)C(C(O)C(O)C5CO)O)C1OC(=C2c(c4)ccc(c4)O)C(c(c3O)c(cc(c3)O)O2)=O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
69 75 0 0 0 0 0 0 0 0999 V2000
-3.7607 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7607 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0461 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3316 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3316 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0461 3.7076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6171 2.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9026 2.4701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9026 3.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6171 3.7076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6171 1.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0030 3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7253 3.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4535 3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4535 4.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7253 5.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0030 4.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0461 1.2328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2329 5.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4230 3.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0942 2.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9905 2.2168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5064 1.5357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2495 1.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9664 1.8324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4455 2.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7954 2.0104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7282 1.5342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6765 1.4593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0233 0.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3772 0.4081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6665 0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8921 0.0293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6665 -0.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3772 -1.1747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1518 -0.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1744 1.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6126 -0.7945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2432 -1.6747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8324 -1.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4149 -2.2560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2699 -2.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1778 -3.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0638 -2.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4572 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3889 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7992 -2.9121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6194 -2.9121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0295 -3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6194 -4.3328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7992 -4.3328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8490 -3.6224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9406 -1.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4565 -2.1730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1995 -1.8840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9165 -1.8763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3955 -1.3552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7455 -1.6983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7787 -2.1531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7503 -2.2700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2165 -1.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9313 -1.2421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8490 -1.7719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8682 -1.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9505 -2.3632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0403 2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9580 1.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4581 -4.6275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5404 -5.1573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
23 28 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
21 22 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
34 40 1 0 0 0 0
32 28 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 46 1 0 0 0 0
49 52 1 0 0 0 0
53 54 1 1 0 0 0
54 55 1 1 0 0 0
56 55 1 1 0 0 0
56 57 1 0 0 0 0
57 58 1 0 0 0 0
58 53 1 0 0 0 0
54 59 1 0 0 0 0
55 60 1 0 0 0 0
56 61 1 0 0 0 0
39 53 1 0 0 0 0
62 63 1 0 0 0 0
57 62 1 0 0 0 0
64 65 1 0 0 0 0
48 64 1 0 0 0 0
66 67 1 0 0 0 0
26 66 1 0 0 0 0
68 69 1 0 0 0 0
50 68 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 69
M SMT 1 CH2OH
M SBV 1 69 -0.5357 -0.1131
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 64 65
M SBL 2 1 71
M SMT 2 ^OCH3
M SBV 2 71 0.2488 -1.0786
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 66 67
M SBL 3 1 73
M SMT 3 CH2OH
M SBV 3 73 -0.5949 0.0285
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 68 69
M SBL 4 1 75
M SMT 4 ^OCH3
M SBV 4 75 0.8387 0.2948
S SKP 5
ID FL5FAAGA0037
FORMULA C44H50O25
EXACTMASS 978.26411715
AVERAGEMASS 978.8528000000001
SMILES C(C(O1)C(O)C(C(OC(O6)C(O)C(O)C(C(COC(=O)C=Cc(c7)cc(c(O)c7OC)OC)6)OC(O5)C(C(O)C(O)C5CO)O)C1OC(=C2c(c4)ccc(c4)O)C(c(c3O)c(cc(c3)O)O2)=O)O)O
M END