Mol:FL5FA9GL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9516 1.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9516 1.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9516 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9516 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2371 0.6184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2371 0.6184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5227 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5227 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5227 1.8557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5227 1.8557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2372 2.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2372 2.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8083 0.6185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8083 0.6185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0940 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0940 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0939 1.8557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0939 1.8557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8084 2.2682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8084 2.2682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8084 -0.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8084 -0.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6201 2.2680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6201 2.2680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3483 1.8478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3483 1.8478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0763 2.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0763 2.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0764 3.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0764 3.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3483 3.5291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3483 3.5291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6201 3.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6201 3.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2371 -0.2060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2371 -0.2060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9659 -1.7736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9659 -1.7736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3948 -2.5890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3948 -2.5890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3724 -2.3999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3724 -2.3999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3078 -2.7565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3078 -2.7565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8791 -1.9409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8791 -1.9409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9015 -2.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9015 -2.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1420 -1.8748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1420 -1.8748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8404 0.6488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8404 0.6488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0779 0.0090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0779 0.0090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0710 -0.0613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0710 -0.0613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8825 -0.6477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8825 -0.6477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6451 -0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6451 -0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6519 0.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6519 0.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7571 0.5694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7571 0.5694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3623 0.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3623 0.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2300 -0.3735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2300 -0.3735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6366 -0.6706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6366 -0.6706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7750 -1.2345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7750 -1.2345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8817 -2.3654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8817 -2.3654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8911 -3.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8911 -3.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3003 -3.5291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3003 -3.5291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1581 0.8878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1581 0.8878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5878 2.3038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5878 2.3038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2300 3.4160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2300 3.4160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 23 36 1 0 0 0 0 | + | 23 36 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 20 37 1 0 0 0 0 | + | 20 37 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 32 8 1 0 0 0 0 | + | 32 8 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 46 0.6363 -0.4480 | + | M SBV 1 46 0.6363 -0.4480 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FA9GL0003 | + | ID FL5FA9GL0003 |
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
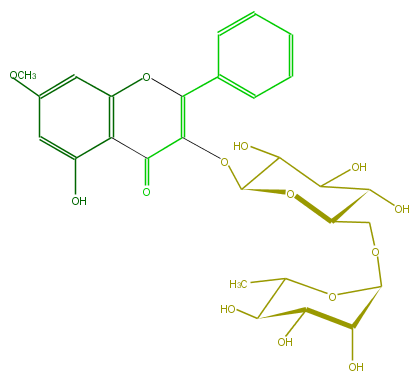
| − | SMILES O(C1OC(=C4c(c5)cccc5)C(c(c3O)c(O4)cc(c3)OC)=O)C(COC(O2)C(C(C(O)C(C)2)O)O)C(C(C1O)O)O | + | SMILES O(C1OC(=C4c(c5)cccc5)C(c(c3O)c(O4)cc(c3)OC)=O)C(COC(O2)C(C(C(O)C(C)2)O)O)C(C(C1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.9516 1.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9516 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2371 0.6184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5227 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5227 1.8557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2372 2.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8083 0.6185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0940 1.0309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0939 1.8557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8084 2.2682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8084 -0.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6201 2.2680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3483 1.8478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0763 2.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0764 3.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3483 3.5291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6201 3.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2371 -0.2060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9659 -1.7736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3948 -2.5890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3724 -2.3999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3078 -2.7565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8791 -1.9409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9015 -2.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1420 -1.8748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8404 0.6488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0779 0.0090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0710 -0.0613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8825 -0.6477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6451 -0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6519 0.0626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7571 0.5694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3623 0.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2300 -0.3735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6366 -0.6706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7750 -1.2345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8817 -2.3654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8911 -3.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3003 -3.5291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1581 0.8878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5878 2.3038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2300 3.4160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
31 33 1 0 0 0 0
30 34 1 0 0 0 0
29 35 1 0 0 0 0
23 36 1 0 0 0 0
35 36 1 0 0 0 0
20 37 1 0 0 0 0
21 38 1 0 0 0 0
22 39 1 0 0 0 0
32 8 1 0 0 0 0
27 32 1 0 0 0 0
26 40 1 0 0 0 0
41 42 1 0 0 0 0
1 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ OCH3
M SBV 1 46 0.6363 -0.4480
S SKP 5
ID FL5FA9GL0003
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES O(C1OC(=C4c(c5)cccc5)C(c(c3O)c(O4)cc(c3)OC)=O)C(COC(O2)C(C(C(O)C(C)2)O)O)C(C(C1O)O)O
M END