Mol:FL4DRNNR0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 48 0 0 0 0 0 0 0 0999 V2000 | + | 42 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3905 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3905 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1304 -0.0543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1304 -0.0543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6512 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6512 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6512 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6512 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1304 1.1486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1304 1.1486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3905 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3905 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1721 -0.0543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1721 -0.0543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6930 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6930 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6930 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6930 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1721 1.1486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1721 1.1486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1721 -0.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1721 -0.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1304 -0.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1304 -0.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8972 1.1404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8972 1.1404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2650 0.0605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2650 0.0605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6186 0.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6186 0.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2650 1.0337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2650 1.0337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2167 0.6100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2167 0.6100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4614 1.1595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4614 1.1595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1078 1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1078 1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5097 1.5832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5097 1.5832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0590 1.2223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0590 1.2223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7558 1.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7558 1.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7558 -0.3512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7558 -0.3512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2969 -0.6975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2969 -0.6975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2969 -1.3299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2969 -1.3299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7493 -1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7493 -1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8445 -1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8445 -1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9109 -0.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9109 -0.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4412 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4412 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9715 -0.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9715 -0.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9715 -0.6664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9715 -0.6664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4412 -0.9725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4412 -0.9725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9109 -0.6664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9109 -0.6664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5566 -0.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5566 -0.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1226 -1.3660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1226 -1.3660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0577 -0.1025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0577 -0.1025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5588 -0.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5588 -0.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5588 -0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5588 -0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0577 -1.2598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0577 -1.2598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5566 -0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5566 -0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0590 -1.2592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0590 -1.2592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1707 -1.4411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1707 -1.4411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 2 12 1 0 0 0 0 | + | 2 12 1 0 0 0 0 |
| − | 6 13 1 0 0 0 0 | + | 6 13 1 0 0 0 0 |
| − | 8 14 1 0 0 0 0 | + | 8 14 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 9 1 0 0 0 0 | + | 16 9 1 0 0 0 0 |
| − | 15 17 1 0 0 0 0 | + | 15 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 16 1 0 0 0 0 | + | 20 16 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 9 22 1 0 0 0 0 | + | 9 22 1 0 0 0 0 |
| − | 8 23 1 0 0 0 0 | + | 8 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 2 0 0 0 0 | + | 33 28 2 0 0 0 0 |
| − | 30 34 1 6 0 0 0 | + | 30 34 1 6 0 0 0 |
| − | 32 35 1 6 0 0 0 | + | 32 35 1 6 0 0 0 |
| − | 34 36 2 0 0 0 0 | + | 34 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 34 1 0 0 0 0 | + | 40 34 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 32 42 1 0 0 0 0 | + | 32 42 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL4DRNNR0001 | + | ID FL4DRNNR0001 |
| − | KNApSAcK_ID C00008665 | + | KNApSAcK_ID C00008665 |
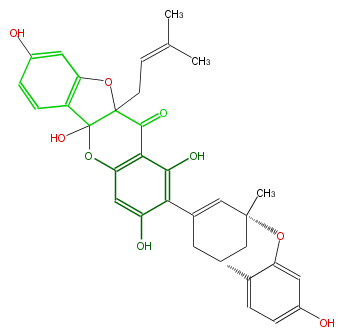
| − | NAME Sanggenon B | + | NAME Sanggenon B |
| − | CAS_RN 81381-67-1 | + | CAS_RN 81381-67-1 |
| − | FORMULA C33H30O9 | + | FORMULA C33H30O9 |
| − | EXACTMASS 570.188982558 | + | EXACTMASS 570.188982558 |
| − | AVERAGEMASS 570.5859 | + | AVERAGEMASS 570.5859 |
| − | SMILES c(c(C(=C6)CC(C7)c(c5)c(OC6(C)7)cc(c5)O)1)(O)cc(O2)c(C(C(O3)(C2(O)c(c4)c3cc(c4)O)CC=C(C)C)=O)c1O | + | SMILES c(c(C(=C6)CC(C7)c(c5)c(OC6(C)7)cc(c5)O)1)(O)cc(O2)c(C(C(O3)(C2(O)c(c4)c3cc(c4)O)CC=C(C)C)=O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 48 0 0 0 0 0 0 0 0999 V2000
-0.3905 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1304 -0.0543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6512 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6512 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1304 1.1486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3905 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1721 -0.0543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6930 0.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6930 0.8479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1721 1.1486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1721 -0.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1304 -0.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8972 1.1404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2650 0.0605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6186 0.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2650 1.0337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2167 0.6100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4614 1.1595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1078 1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5097 1.5832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0590 1.2223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7558 1.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7558 -0.3512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2969 -0.6975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2969 -1.3299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7493 -1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8445 -1.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9109 -0.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4412 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9715 -0.0540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9715 -0.6664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4412 -0.9725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9109 -0.6664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5566 -0.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1226 -1.3660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0577 -0.1025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5588 -0.3919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5588 -0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0577 -1.2598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5566 -0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0590 -1.2592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1707 -1.4411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
2 12 1 0 0 0 0
6 13 1 0 0 0 0
8 14 1 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 9 1 0 0 0 0
15 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 16 1 0 0 0 0
18 21 1 0 0 0 0
9 22 1 0 0 0 0
8 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
25 27 1 0 0 0 0
1 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 28 2 0 0 0 0
30 34 1 6 0 0 0
32 35 1 6 0 0 0
34 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 34 1 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
32 42 1 0 0 0 0
S SKP 8
ID FL4DRNNR0001
KNApSAcK_ID C00008665
NAME Sanggenon B
CAS_RN 81381-67-1
FORMULA C33H30O9
EXACTMASS 570.188982558
AVERAGEMASS 570.5859
SMILES c(c(C(=C6)CC(C7)c(c5)c(OC6(C)7)cc(c5)O)1)(O)cc(O2)c(C(C(O3)(C2(O)c(c4)c3cc(c4)O)CC=C(C)C)=O)c1O
M END