Mol:FL4DACGS0020
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.2998 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2998 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7789 -0.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7789 -0.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2580 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2580 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2580 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2580 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7789 0.5203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7789 0.5203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2998 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2998 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7371 -0.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7371 -0.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2162 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2162 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2162 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2162 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7371 0.5203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7371 0.5203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7371 -1.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7371 -1.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6365 0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6365 0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1078 0.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1078 0.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5791 0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5791 0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5791 1.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5791 1.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1078 1.4348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1078 1.4348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6365 1.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6365 1.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7504 0.5068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7504 0.5068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7480 -0.9586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7480 -0.9586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0504 1.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0504 1.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7789 -1.2835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7789 -1.2835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9782 0.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9782 0.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6774 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6774 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2559 0.3452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2559 0.3452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8379 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8379 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1388 0.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1388 0.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5602 0.5357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5602 0.5357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4196 0.5512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4196 0.5512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7984 0.9482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7984 0.9482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5728 0.4503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5728 0.4503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0018 0.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0018 0.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4020 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4020 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6926 -0.3233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6926 -0.3233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6926 -0.9433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6926 -0.9433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1693 -1.4200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1693 -1.4200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6598 -0.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6598 -0.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2011 -1.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2011 -1.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2051 -1.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2051 -1.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7504 -0.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7504 -0.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7504 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7504 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2051 0.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2051 0.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6598 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6598 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 31 14 1 0 0 0 0 | + | 31 14 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 34 37 2 0 0 0 0 | + | 34 37 2 0 0 0 0 |
| − | 36 38 2 0 0 0 0 | + | 36 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 36 1 0 0 0 0 | + | 42 36 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL4DACGS0020 | + | ID FL4DACGS0020 |
| − | KNApSAcK_ID C00008712 | + | KNApSAcK_ID C00008712 |
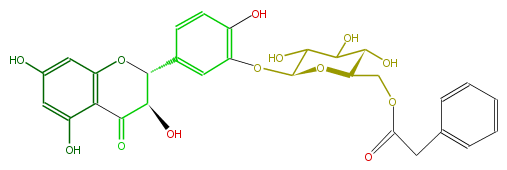
| − | NAME Taxifolin 3'-(6''-phenylacetylglucoside) | + | NAME Taxifolin 3'-(6''-phenylacetylglucoside) |
| − | CAS_RN 95519-27-0 | + | CAS_RN 95519-27-0 |
| − | FORMULA C29H28O13 | + | FORMULA C29H28O13 |
| − | EXACTMASS 584.152990982 | + | EXACTMASS 584.152990982 |
| − | AVERAGEMASS 584.52482 | + | AVERAGEMASS 584.52482 |
| − | SMILES c(c1)(O)cc(c(C(=O)2)c1OC(c(c3)ccc(O)c(OC(O4)C(O)C(C(C(COC(Cc(c5)cccc5)=O)4)O)O)3)C2O)O | + | SMILES c(c1)(O)cc(c(C(=O)2)c1OC(c(c3)ccc(O)c(OC(O4)C(O)C(C(C(COC(Cc(c5)cccc5)=O)4)O)O)3)C2O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-4.2998 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7789 -0.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2580 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2580 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7789 0.5203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2998 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7371 -0.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2162 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2162 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7371 0.5203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7371 -1.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6365 0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1078 0.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5791 0.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5791 1.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1078 1.4348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6365 1.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7504 0.5068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7480 -0.9586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0504 1.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7789 -1.2835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9782 0.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6774 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2559 0.3452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8379 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1388 0.7009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5602 0.5357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4196 0.5512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7984 0.9482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5728 0.4503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0018 0.3619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4020 0.1799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6926 -0.3233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6926 -0.9433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1693 -1.4200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6598 -0.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2011 -1.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2051 -1.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7504 -0.9295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7504 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2051 0.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6598 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 31 1 0 0 0 0
31 14 1 0 0 0 0
25 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
34 37 2 0 0 0 0
36 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 36 1 0 0 0 0
S SKP 8
ID FL4DACGS0020
KNApSAcK_ID C00008712
NAME Taxifolin 3'-(6''-phenylacetylglucoside)
CAS_RN 95519-27-0
FORMULA C29H28O13
EXACTMASS 584.152990982
AVERAGEMASS 584.52482
SMILES c(c1)(O)cc(c(C(=O)2)c1OC(c(c3)ccc(O)c(OC(O4)C(O)C(C(C(COC(Cc(c5)cccc5)=O)4)O)O)3)C2O)O
M END