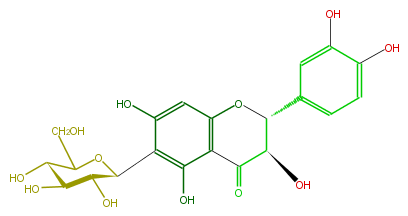
Mol:FL4DACCS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9848 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9848 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9848 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9848 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4285 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4285 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1278 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1278 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1278 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1278 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4285 0.0959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4285 0.0959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6841 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6841 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2404 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2404 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2404 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2404 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6841 0.0959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6841 0.0959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6841 -1.6897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6841 -1.6897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4285 -1.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4285 -1.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9203 0.1989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9203 0.1989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5065 -0.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5065 -0.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0927 0.1989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0927 0.1989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0927 0.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0927 0.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5065 1.2142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5065 1.2142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9203 0.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9203 0.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6786 1.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6786 1.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1647 -1.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1647 -1.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7936 -1.5845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7936 -1.5845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2591 -1.3766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2591 -1.3766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7433 -1.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7433 -1.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1181 -0.9962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1181 -0.9962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5857 -1.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5857 -1.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6786 -1.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6786 -1.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3722 -1.6127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3722 -1.6127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9528 -1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9528 -1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5065 1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5065 1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9105 -1.5377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9105 -1.5377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5409 0.0958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5409 0.0958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9841 -0.4588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9841 -0.4588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1873 -0.6723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1873 -0.6723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 17 29 1 0 0 0 0 | + | 17 29 1 0 0 0 0 |
| − | 8 30 1 1 0 0 0 | + | 8 30 1 1 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 35 -6.4322 7.1589 | + | M SBV 1 35 -6.4322 7.1589 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL4DACCS0001 | + | ID FL4DACCS0001 |
| − | KNApSAcK_ID C00006095 | + | KNApSAcK_ID C00006095 |
| − | NAME 6-C-Glucopyranosyldihydroquercetin | + | NAME 6-C-Glucopyranosyldihydroquercetin |
| − | CAS_RN 112494-39-0 | + | CAS_RN 112494-39-0 |
| − | FORMULA C21H22O12 | + | FORMULA C21H22O12 |
| − | EXACTMASS 466.111126168 | + | EXACTMASS 466.111126168 |
| − | AVERAGEMASS 466.39218 | + | AVERAGEMASS 466.39218 |
| − | SMILES O(C(c(c4)cc(c(O)c4)O)3)c(c(C(C(O)3)=O)2)cc(c(c2O)C(C(O)1)OC(CO)C(O)C1O)O | + | SMILES O(C(c(c4)cc(c(O)c4)O)3)c(c(C(C(O)3)=O)2)cc(c(c2O)C(C(O)1)OC(CO)C(O)C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-0.9848 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9848 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4285 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1278 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1278 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4285 0.0959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6841 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2404 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2404 -0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6841 0.0959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6841 -1.6897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4285 -1.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9203 0.1989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5065 -0.1396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0927 0.1989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0927 0.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5065 1.2142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9203 0.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6786 1.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1647 -1.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7936 -1.5845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2591 -1.3766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7433 -1.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1181 -0.9962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5857 -1.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6786 -1.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3722 -1.6127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9528 -1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5065 1.8907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9105 -1.5377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5409 0.0958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9841 -0.4588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1873 -0.6723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 6 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
2 23 1 0 0 0 0
17 29 1 0 0 0 0
8 30 1 1 0 0 0
1 31 1 0 0 0 0
25 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 ^CH2OH
M SBV 1 35 -6.4322 7.1589
S SKP 8
ID FL4DACCS0001
KNApSAcK_ID C00006095
NAME 6-C-Glucopyranosyldihydroquercetin
CAS_RN 112494-39-0
FORMULA C21H22O12
EXACTMASS 466.111126168
AVERAGEMASS 466.39218
SMILES O(C(c(c4)cc(c(O)c4)O)3)c(c(C(C(O)3)=O)2)cc(c(c2O)C(C(O)1)OC(CO)C(O)C1O)O
M END