Mol:FL3FGCGS0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 50 0 0 0 0 0 0 0 0999 V2000 | + | 47 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.6977 -0.1247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6977 -0.1247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6977 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6977 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2467 -0.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2467 -0.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7956 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7956 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7956 -0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7956 -0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2467 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2467 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3445 -0.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3445 -0.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8935 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8935 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8935 -0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8935 -0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3445 0.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3445 0.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1728 -1.3151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1728 -1.3151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4426 0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4426 0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9828 -0.1710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9828 -0.1710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5231 0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5231 0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5231 0.6252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5231 0.6252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9828 0.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9828 0.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4426 0.6252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4426 0.6252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2467 -1.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2467 -1.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1506 0.1742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1506 0.1742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4769 0.6252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4769 0.6252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2771 1.6798 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.2771 1.6798 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.8895 1.0084 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8895 1.0084 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6349 1.2215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6349 1.2215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3849 1.0084 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3849 1.0084 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7726 1.6798 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7726 1.6798 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.0271 1.4669 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.0271 1.4669 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5572 1.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5572 1.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3341 1.9985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3341 1.9985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2293 2.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2293 2.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9672 0.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9672 0.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7396 -0.0245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7396 -0.0245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3201 -0.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3201 -0.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6601 -0.2347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6601 -0.2347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2730 -0.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2730 -0.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7262 -1.2238 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7262 -1.2238 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.6715 -1.8492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6715 -1.8492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1484 -2.0930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1484 -2.0930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5785 -1.6940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5785 -1.6940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0497 -2.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0497 -2.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3289 -0.9456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3289 -0.9456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1090 -1.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1090 -1.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4122 -0.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4122 -0.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3956 -0.3731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3956 -0.3731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0078 0.6449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0078 0.6449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6096 1.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6096 1.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8146 1.4718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8146 1.4718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5366 2.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5366 2.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 2 42 1 0 0 0 0 | + | 2 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 6 44 1 0 0 0 0 | + | 6 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 16 46 1 0 0 0 0 | + | 16 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 46 47 | + | M SAL 4 2 46 47 |
| − | M SBL 4 1 49 | + | M SBL 4 1 49 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 49 -0.8146 1.4718 | + | M SVB 4 49 -0.8146 1.4718 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 44 45 | + | M SAL 3 2 44 45 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 47 -3.0078 0.6449 | + | M SVB 3 47 -3.0078 0.6449 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 45 -4.4122 -0.5548 | + | M SVB 2 45 -4.4122 -0.5548 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 37 38 39 | + | M SAL 1 3 37 38 39 |
| − | M SBL 1 1 40 | + | M SBL 1 1 40 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 40 3.9701 -1.6678 | + | M SVB 1 40 3.9701 -1.6678 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FGCGS0009 | + | ID FL3FGCGS0009 |
| − | KNApSAcK_ID C00004447 | + | KNApSAcK_ID C00004447 |
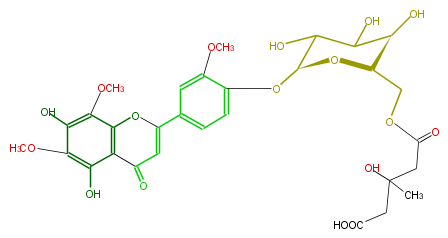
| − | NAME 5,7,4'-Trihydroxy-6,8,3'-trimethoxyflavone 4'-[6''-(3-hydroxy-3-methylglutaryl)glucoside] | + | NAME 5,7,4'-Trihydroxy-6,8,3'-trimethoxyflavone 4'-[6''-(3-hydroxy-3-methylglutaryl)glucoside] |
| − | CAS_RN 98891-96-4 | + | CAS_RN 98891-96-4 |
| − | FORMULA C30H34O17 | + | FORMULA C30H34O17 |
| − | EXACTMASS 666.179599662 | + | EXACTMASS 666.179599662 |
| − | AVERAGEMASS 666.58076 | + | AVERAGEMASS 666.58076 |
| − | SMILES c(c21)(OC(c(c4)ccc(c(OC)4)O[C@H](O3)C(C([C@@H](O)[C@H]3COC(CC(CC(O)=O)(C)O)=O)O)O)=CC2=O)c(OC)c(c(OC)c1O)O | + | SMILES c(c21)(OC(c(c4)ccc(c(OC)4)O[C@H](O3)C(C([C@@H](O)[C@H]3COC(CC(CC(O)=O)(C)O)=O)O)O)=CC2=O)c(OC)c(c(OC)c1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 50 0 0 0 0 0 0 0 0999 V2000
-3.6977 -0.1247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6977 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2467 -0.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7956 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7956 -0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2467 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3445 -0.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8935 -0.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8935 -0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3445 0.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1728 -1.3151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4426 0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9828 -0.1710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5231 0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5231 0.6252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9828 0.8907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4426 0.6252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2467 -1.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1506 0.1742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4769 0.6252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2771 1.6798 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.8895 1.0084 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6349 1.2215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3849 1.0084 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7726 1.6798 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.0271 1.4669 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5572 1.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3341 1.9985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2293 2.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9672 0.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7396 -0.0245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3201 -0.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6601 -0.2347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2730 -0.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7262 -1.2238 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.6715 -1.8492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1484 -2.0930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5785 -1.6940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0497 -2.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3289 -0.9456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1090 -1.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4122 -0.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3956 -0.3731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0078 0.6449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6096 1.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8146 1.4718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5366 2.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
37 39 1 0 0 0 0
35 40 1 0 0 0 0
35 41 1 0 0 0 0
2 42 1 0 0 0 0
42 43 1 0 0 0 0
6 44 1 0 0 0 0
44 45 1 0 0 0 0
16 46 1 0 0 0 0
46 47 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 46 47
M SBL 4 1 49
M SMT 4 OCH3
M SVB 4 49 -0.8146 1.4718
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 44 45
M SBL 3 1 47
M SMT 3 OCH3
M SVB 3 47 -3.0078 0.6449
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 45
M SMT 2 OCH3
M SVB 2 45 -4.4122 -0.5548
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 37 38 39
M SBL 1 1 40
M SMT 1 COOH
M SVB 1 40 3.9701 -1.6678
S SKP 8
ID FL3FGCGS0009
KNApSAcK_ID C00004447
NAME 5,7,4'-Trihydroxy-6,8,3'-trimethoxyflavone 4'-[6''-(3-hydroxy-3-methylglutaryl)glucoside]
CAS_RN 98891-96-4
FORMULA C30H34O17
EXACTMASS 666.179599662
AVERAGEMASS 666.58076
SMILES c(c21)(OC(c(c4)ccc(c(OC)4)O[C@H](O3)C(C([C@@H](O)[C@H]3COC(CC(CC(O)=O)(C)O)=O)O)O)=CC2=O)c(OC)c(c(OC)c1O)O
M END