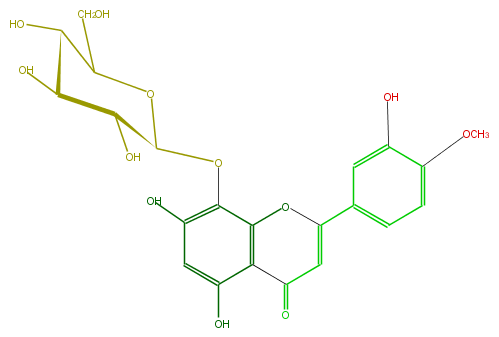
Mol:FL3FFCGS0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7869 -1.1247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7869 -1.1247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7869 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7869 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0861 -2.4029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0861 -2.4029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3851 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3851 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3851 -1.1887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3851 -1.1887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0861 -0.7841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0861 -0.7841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3159 -2.4029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3159 -2.4029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0168 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0168 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0168 -1.1887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0168 -1.1887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3159 -0.7841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3159 -0.7841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3159 -3.0339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3159 -3.0339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7175 -0.7842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7175 -0.7842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4320 -1.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4320 -1.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1463 -0.7842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1463 -0.7842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1463 0.0406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1463 0.0406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4320 0.4532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4320 0.4532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7175 0.0406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7175 0.0406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0861 -3.2105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0861 -3.2105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4907 -0.6602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4907 -0.6602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0861 0.1147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0861 0.1147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4130 1.4791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4130 1.4791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3462 2.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3462 2.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4123 1.5168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4123 1.5168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2399 1.1188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2399 1.1188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3794 0.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3794 0.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4790 1.5522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4790 1.5522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6538 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6538 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1778 2.9997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1778 2.9997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1387 2.0449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1387 2.0449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9298 0.2341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9298 0.2341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0675 0.7083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0675 0.7083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1778 0.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1778 0.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9229 3.2105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9229 3.2105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8127 2.5694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8127 2.5694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 15 31 1 0 0 0 0 | + | 15 31 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 35 -0.9212 -0.6677 | + | M SBV 1 35 -0.9212 -0.6677 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 33 34 | + | M SAL 2 2 33 34 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 37 0.2691 -1.2248 | + | M SBV 2 37 0.2691 -1.2248 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FFCGS0015 | + | ID FL3FFCGS0015 |
| − | FORMULA C22H22O12 | + | FORMULA C22H22O12 |
| − | EXACTMASS 478.111126168 | + | EXACTMASS 478.111126168 |
| − | AVERAGEMASS 478.40288000000004 | + | AVERAGEMASS 478.40288000000004 |
| − | SMILES Oc(c24)cc(c(c(OC(=CC4=O)c(c3)ccc(c3O)OC)2)OC(O1)C(O)C(C(C(CO)1)O)O)O | + | SMILES Oc(c24)cc(c(c(OC(=CC4=O)c(c3)ccc(c3O)OC)2)OC(O1)C(O)C(C(C(CO)1)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-1.7869 -1.1247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7869 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0861 -2.4029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3851 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3851 -1.1887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0861 -0.7841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3159 -2.4029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0168 -1.9982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0168 -1.1887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3159 -0.7841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3159 -3.0339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7175 -0.7842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4320 -1.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1463 -0.7842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1463 0.0406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4320 0.4532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7175 0.0406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0861 -3.2105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4907 -0.6602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0861 0.1147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4130 1.4791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3462 2.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4123 1.5168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2399 1.1188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3794 0.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4790 1.5522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6538 1.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1778 2.9997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1387 2.0449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9298 0.2341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0675 0.7083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1778 0.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9229 3.2105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8127 2.5694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
6 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 20 1 0 0 0 0
31 32 1 0 0 0 0
15 31 1 0 0 0 0
33 34 1 0 0 0 0
27 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 35
M SMT 1 OCH3
M SBV 1 35 -0.9212 -0.6677
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 33 34
M SBL 2 1 37
M SMT 2 ^CH2OH
M SBV 2 37 0.2691 -1.2248
S SKP 5
ID FL3FFCGS0015
FORMULA C22H22O12
EXACTMASS 478.111126168
AVERAGEMASS 478.40288000000004
SMILES Oc(c24)cc(c(c(OC(=CC4=O)c(c3)ccc(c3O)OC)2)OC(O1)C(O)C(C(C(CO)1)O)O)O
M END