Mol:FL3FFAGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.4240 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4240 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4240 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4240 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8750 0.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8750 0.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3261 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3261 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3261 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3261 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8750 1.1777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8750 1.1777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7772 0.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7772 0.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2282 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2282 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2282 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2282 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7772 1.1777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7772 1.1777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7772 -0.2701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7772 -0.2701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6791 1.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6791 1.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1389 0.9122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1389 0.9122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5986 1.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5986 1.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5986 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5986 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1389 1.9738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1389 1.9738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6791 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6791 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8750 -0.2704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8750 -0.2704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3327 2.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3327 2.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0955 1.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0955 1.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8750 1.7735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8750 1.7735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9106 1.1044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9106 1.1044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3950 0.4238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3950 0.4238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6525 0.7125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6525 0.7125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9360 0.7202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9360 0.7202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4567 1.2410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4567 1.2410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2151 0.9686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2151 0.9686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4871 0.7715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4871 0.7715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1988 0.3846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1988 0.3846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2271 -0.0017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2271 -0.0017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7562 -0.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7562 -0.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2406 -1.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2406 -1.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4981 -1.2262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4981 -1.2262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7817 -1.2185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7817 -1.2185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3023 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3023 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0607 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0607 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3327 -1.1672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3327 -1.1672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1097 -2.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1097 -2.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9792 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9792 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5107 1.7760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5107 1.7760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7962 1.3635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7962 1.3635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3563 -0.1627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3563 -0.1627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6418 -0.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6418 -0.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 3 1 0 0 0 0 | + | 18 3 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 44 -7.3707 6.6229 | + | M SBV 1 44 -7.3707 6.6229 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 46 -7.3707 6.6229 | + | M SBV 2 46 -7.3707 6.6229 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FFAGS0004 | + | ID FL3FFAGS0004 |
| − | KNApSAcK_ID C00004248 | + | KNApSAcK_ID C00004248 |
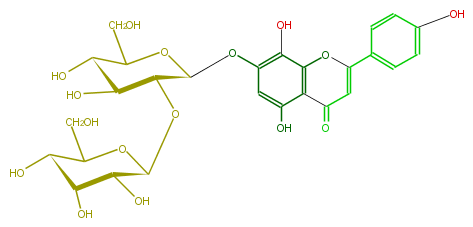
| − | NAME Isoscutellarein 7-allosyl-(1->2)-glucoside | + | NAME Isoscutellarein 7-allosyl-(1->2)-glucoside |
| − | CAS_RN 96627-12-2 | + | CAS_RN 96627-12-2 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(O5)(=CC(c(c52)c(O)cc(OC(C(OC(C4O)OC(C(C4O)O)CO)3)OC(C(C3O)O)CO)c2O)=O)c(c1)ccc(O)c1 | + | SMILES C(O5)(=CC(c(c52)c(O)cc(OC(C(OC(C4O)OC(C(C4O)O)CO)3)OC(C(C3O)O)CO)c2O)=O)c(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.4240 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4240 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8750 0.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3261 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3261 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8750 1.1777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7772 0.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2282 0.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2282 0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7772 1.1777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7772 -0.2701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6791 1.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1389 0.9122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5986 1.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5986 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1389 1.9738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6791 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8750 -0.2704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3327 2.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0955 1.2172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8750 1.7735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9106 1.1044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3950 0.4238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6525 0.7125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9360 0.7202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4567 1.2410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2151 0.9686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4871 0.7715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1988 0.3846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2271 -0.0017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7562 -0.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2406 -1.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4981 -1.2262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7817 -1.2185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3023 -0.6978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0607 -0.9701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3327 -1.1672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1097 -2.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9792 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5107 1.7760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7962 1.3635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3563 -0.1627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6418 -0.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 3 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
6 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
34 30 1 0 0 0 0
33 39 1 0 0 0 0
27 40 1 0 0 0 0
40 41 1 0 0 0 0
36 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 ^CH2OH
M SBV 1 44 -7.3707 6.6229
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 ^CH2OH
M SBV 2 46 -7.3707 6.6229
S SKP 8
ID FL3FFAGS0004
KNApSAcK_ID C00004248
NAME Isoscutellarein 7-allosyl-(1->2)-glucoside
CAS_RN 96627-12-2
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(O5)(=CC(c(c52)c(O)cc(OC(C(OC(C4O)OC(C(C4O)O)CO)3)OC(C(C3O)O)CO)c2O)=O)c(c1)ccc(O)c1
M END