Mol:FL3FECGS0040
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 43 0 0 0 0 0 0 0 0999 V2000 | + | 40 43 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1265 0.4071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1265 0.4071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6694 0.2616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6694 0.2616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0377 0.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0377 0.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9029 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9029 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3998 1.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3998 1.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0316 0.8995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0316 0.8995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2712 1.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2712 1.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1364 2.0044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1364 2.0044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6333 2.1391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6333 2.1391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2651 1.7709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2651 1.7709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6710 1.5719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6710 1.5719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4986 2.6421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4986 2.6421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8739 3.0175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8739 3.0175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7366 3.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7366 3.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2238 3.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2238 3.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1516 3.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1516 3.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0142 2.7795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0142 2.7795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5398 0.4953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5398 0.4953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2502 0.0305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2502 0.0305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0864 4.1803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0864 4.1803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7920 -0.3061 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.7920 -0.3061 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.2763 -0.9867 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2763 -0.9867 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5338 -0.6980 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5338 -0.6980 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.8174 -0.6903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.8174 -0.6903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3380 -0.1695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3380 -0.1695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9876 -0.5124 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.9876 -0.5124 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.5057 0.1060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5057 0.1060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8451 -1.6933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8451 -1.6933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1084 -1.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1084 -1.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1026 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1026 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6643 3.4297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6643 3.4297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7203 0.6859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7203 0.6859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9876 1.2591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9876 1.2591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6163 1.3140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6163 1.3140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6256 1.7760 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.6256 1.7760 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.8923 2.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8923 2.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9969 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9969 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5211 2.4030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5211 2.4030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9711 -0.3992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9711 -0.3992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3860 -1.3088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3860 -1.3088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 2 39 1 0 0 0 0 | + | 2 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 42 | + | M SBL 1 1 42 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 42 -0.2519 -0.9732 | + | M SVB 1 42 -0.2519 -0.9732 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FECGS0040 | + | ID FL3FECGS0040 |
| − | KNApSAcK_ID C00004516 | + | KNApSAcK_ID C00004516 |
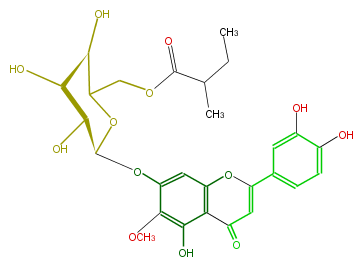
| − | NAME 6-Methoxyluteolin 7-[6''-(2-methylbutyryl) glucoside];2-(3,4-Dihydroxyphenyl)-5-hydroxy-6-methoxy-7-[[6-O-(2-methyl-1-oxobutyl)-beta-D-glucopyranosyl]oxy]-4H-1-benzopyran-4-one | + | NAME 6-Methoxyluteolin 7-[6''-(2-methylbutyryl) glucoside];2-(3,4-Dihydroxyphenyl)-5-hydroxy-6-methoxy-7-[[6-O-(2-methyl-1-oxobutyl)-beta-D-glucopyranosyl]oxy]-4H-1-benzopyran-4-one |
| − | CAS_RN 144599-15-5 | + | CAS_RN 144599-15-5 |
| − | FORMULA C27H30O13 | + | FORMULA C27H30O13 |
| − | EXACTMASS 562.168641046 | + | EXACTMASS 562.168641046 |
| − | AVERAGEMASS 562.5193 | + | AVERAGEMASS 562.5193 |
| − | SMILES O=C(C=1)c(c4O)c(cc(c(OC)4)O[C@H](O3)[C@H]([C@H]([C@@H](O)C(COC(=O)C(CC)C)3)O)O)OC1c(c2)ccc(O)c2O | + | SMILES O=C(C=1)c(c4O)c(cc(c(OC)4)O[C@H](O3)[C@H]([C@H]([C@@H](O)C(COC(=O)C(CC)C)3)O)O)OC1c(c2)ccc(O)c2O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 43 0 0 0 0 0 0 0 0999 V2000
0.1265 0.4071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6694 0.2616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0377 0.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9029 1.1330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3998 1.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0316 0.8995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2712 1.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1364 2.0044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6333 2.1391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2651 1.7709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6710 1.5719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4986 2.6421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8739 3.0175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7366 3.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2238 3.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1516 3.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0142 2.7795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5398 0.4953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2502 0.0305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0864 4.1803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7920 -0.3061 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.2763 -0.9867 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5338 -0.6980 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8174 -0.6903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3380 -0.1695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9876 -0.5124 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.5057 0.1060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8451 -1.6933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1084 -1.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1026 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6643 3.4297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7203 0.6859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9876 1.2591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6163 1.3140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6256 1.7760 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.8923 2.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9969 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5211 2.4030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9711 -0.3992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3860 -1.3088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 19 1 0 0 0 0
26 30 1 0 0 0 0
16 31 1 0 0 0 0
30 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 1 0 0 0 0
35 37 1 0 0 0 0
36 38 1 0 0 0 0
2 39 1 0 0 0 0
39 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 42
M SMT 1 OCH3
M SVB 1 42 -0.2519 -0.9732
S SKP 8
ID FL3FECGS0040
KNApSAcK_ID C00004516
NAME 6-Methoxyluteolin 7-[6''-(2-methylbutyryl) glucoside];2-(3,4-Dihydroxyphenyl)-5-hydroxy-6-methoxy-7-[[6-O-(2-methyl-1-oxobutyl)-beta-D-glucopyranosyl]oxy]-4H-1-benzopyran-4-one
CAS_RN 144599-15-5
FORMULA C27H30O13
EXACTMASS 562.168641046
AVERAGEMASS 562.5193
SMILES O=C(C=1)c(c4O)c(cc(c(OC)4)O[C@H](O3)[C@H]([C@H]([C@@H](O)C(COC(=O)C(CC)C)3)O)O)OC1c(c2)ccc(O)c2O
M END