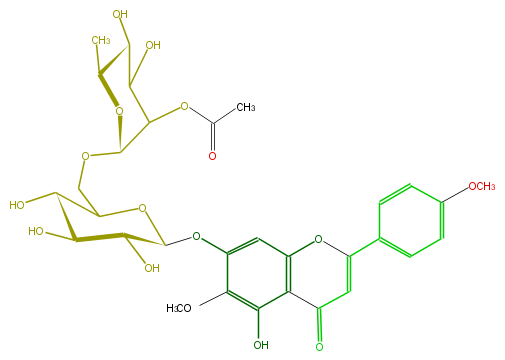
Mol:FL3FEAGS0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 51 0 0 0 0 0 0 0 0999 V2000 | + | 47 51 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9425 -1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9425 -1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9425 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9425 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2805 -2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2805 -2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3815 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3815 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3815 -1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3815 -1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2805 -1.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2805 -1.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0436 -2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0436 -2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7056 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7056 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7056 -1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7056 -1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0436 -1.2402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0436 -1.2402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0594 -3.5902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0594 -3.5902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3673 -1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3673 -1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0421 -1.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0421 -1.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7168 -1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7168 -1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7168 -0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7168 -0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0421 -0.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0421 -0.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3673 -0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3673 -0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2805 -3.5319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2805 -3.5319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6196 -1.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6196 -1.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6786 -0.2258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6786 -0.2258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2534 -1.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2534 -1.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2074 -1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2074 -1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2904 -1.3912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2904 -1.3912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7767 -0.5486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7767 -0.5486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7228 -0.7641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7228 -0.7641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1947 -0.1445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1947 -0.1445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4401 -0.4884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4401 -0.4884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0229 -1.0677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0229 -1.0677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6583 -1.8649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6583 -1.8649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0286 0.5703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0286 0.5703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0896 2.9796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0896 2.9796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7313 2.3381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7313 2.3381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2818 1.5498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2818 1.5498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2818 0.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2818 0.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6403 1.2842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6403 1.2842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0897 2.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0897 2.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3440 3.6713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3440 3.6713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8180 3.0998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8180 3.0998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6303 2.9848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6303 2.9848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8765 1.6711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8765 1.6711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2609 1.2672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2609 1.2672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2556 0.5762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2556 0.5762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6726 1.6502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6726 1.6502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6606 -2.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6606 -2.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7925 -3.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7925 -3.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3914 -0.0718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3914 -0.0718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4401 -0.6772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4401 -0.6772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 20 27 1 0 0 0 0 | + | 20 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 30 26 1 0 0 0 0 | + | 30 26 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 2 44 1 0 0 0 0 | + | 2 44 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 15 46 1 0 0 0 0 | + | 15 46 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 49 | + | M SBL 1 1 49 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 49 0.7180 0.3502 | + | M SBV 1 49 0.7180 0.3502 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 46 47 | + | M SAL 2 2 46 47 |
| − | M SBL 2 1 51 | + | M SBL 2 1 51 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 51 -0.6746 -0.3895 | + | M SBV 2 51 -0.6746 -0.3895 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0034 | + | ID FL3FEAGS0034 |
| − | FORMULA C31H36O16 | + | FORMULA C31H36O16 |
| − | EXACTMASS 664.200335104 | + | EXACTMASS 664.200335104 |
| − | AVERAGEMASS 664.60794 | + | AVERAGEMASS 664.60794 |
| − | SMILES OC(C(Oc(c(OC)5)cc(c3c(O)5)OC(c(c4)ccc(OC)c4)=CC3=O)2)C(C(O)C(O2)COC(C1OC(C)=O)OC(C)C(O)C(O)1)O | + | SMILES OC(C(Oc(c(OC)5)cc(c3c(O)5)OC(c(c4)ccc(OC)c4)=CC3=O)2)C(C(O)C(O2)COC(C1OC(C)=O)OC(C)C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 51 0 0 0 0 0 0 0 0999 V2000
-0.9425 -1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9425 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2805 -2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3815 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3815 -1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2805 -1.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0436 -2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7056 -2.3869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7056 -1.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0436 -1.2402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0594 -3.5902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3673 -1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0421 -1.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7168 -1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7168 -0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0421 -0.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3673 -0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2805 -3.5319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6196 -1.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6786 -0.2258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2534 -1.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2074 -1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2904 -1.3912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7767 -0.5486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7228 -0.7641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1947 -0.1445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4401 -0.4884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0229 -1.0677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6583 -1.8649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0286 0.5703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0896 2.9796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7313 2.3381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2818 1.5498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2818 0.6426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6403 1.2842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0897 2.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3440 3.6713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8180 3.0998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6303 2.9848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8765 1.6711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2609 1.2672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2556 0.5762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6726 1.6502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6606 -2.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7925 -3.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3914 -0.0718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4401 -0.6772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
25 26 1 0 0 0 0
20 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
30 26 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
34 30 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
23 19 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
44 45 1 0 0 0 0
2 44 1 0 0 0 0
46 47 1 0 0 0 0
15 46 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 49
M SMT 1 ^ OCH3
M SBV 1 49 0.7180 0.3502
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 46 47
M SBL 2 1 51
M SMT 2 OCH3
M SBV 2 51 -0.6746 -0.3895
S SKP 5
ID FL3FEAGS0034
FORMULA C31H36O16
EXACTMASS 664.200335104
AVERAGEMASS 664.60794
SMILES OC(C(Oc(c(OC)5)cc(c3c(O)5)OC(c(c4)ccc(OC)c4)=CC3=O)2)C(C(O)C(O2)COC(C1OC(C)=O)OC(C)C(O)C(O)1)O
M END