Mol:FL3FEAGS0030
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1379 0.4195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1379 0.4195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1379 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1379 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7999 -0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7999 -0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4619 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4619 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4619 0.3590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4619 0.3590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7999 0.7412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7999 0.7412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1240 -0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1240 -0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7860 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7860 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7860 0.3590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7860 0.3590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1240 0.7412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1240 0.7412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1240 -1.5562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1240 -1.5562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4477 0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4477 0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1225 0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1225 0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7973 0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7973 0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7973 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7973 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1225 1.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1225 1.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4477 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4477 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7999 -1.5504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7999 -1.5504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5391 0.8104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5391 0.8104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5752 -0.8172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5752 -0.8172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4719 1.9097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4719 1.9097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6898 0.7620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6898 0.7620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0844 -0.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0844 -0.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2126 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2126 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3713 0.3110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3713 0.3110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9826 0.9224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9826 0.9224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8732 0.6027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8732 0.6027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3668 0.3713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3668 0.3713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0282 -0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0282 -0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4848 -0.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4848 -0.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0631 -1.8048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0631 -1.8048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4101 -1.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4101 -1.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6185 -1.7371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6185 -1.7371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8641 -1.1840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8641 -1.1840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0538 -0.9575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0538 -0.9575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8025 -0.5250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8025 -0.5250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5800 -1.0645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5800 -1.0645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2437 -1.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2437 -1.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8125 -1.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8125 -1.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3969 -0.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3969 -0.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3619 -0.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3619 -0.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4719 -1.1786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4719 -1.1786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3038 1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3038 1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2724 1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2724 1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8177 1.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8177 1.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 2 20 1 0 0 0 0 | + | 2 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 27 43 1 0 0 0 0 | + | 27 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 40 41 42 | + | M SAL 1 3 40 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 46 0.8168 -0.3584 | + | M SBV 1 46 0.8168 -0.3584 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 43 44 45 | + | M SAL 2 3 43 44 45 |
| − | M SBL 2 1 49 | + | M SBL 2 1 49 |
| − | M SMT 2 ^ COOH | + | M SMT 2 ^ COOH |
| − | M SBV 2 49 0.4306 -0.5251 | + | M SBV 2 49 0.4306 -0.5251 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0030 | + | ID FL3FEAGS0030 |
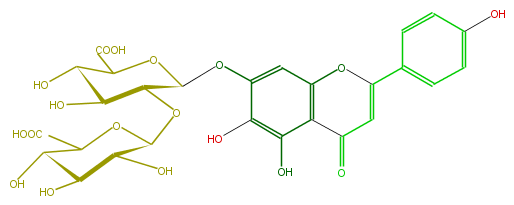
| − | FORMULA C27H26O18 | + | FORMULA C27H26O18 |
| − | EXACTMASS 638.111914028 | + | EXACTMASS 638.111914028 |
| − | AVERAGEMASS 638.4845399999999 | + | AVERAGEMASS 638.4845399999999 |
| − | SMILES c(c1)(ccc(C(O2)=CC(c(c(O)5)c2cc(c(O)5)OC(C3OC(O4)C(O)C(C(C4C(O)=O)O)O)OC(C(C3O)O)C(O)=O)=O)c1)O | + | SMILES c(c1)(ccc(C(O2)=CC(c(c(O)5)c2cc(c(O)5)OC(C3OC(O4)C(O)C(C(C4C(O)=O)O)O)OC(C(C3O)O)C(O)=O)=O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
0.1379 0.4195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1379 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7999 -0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4619 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4619 0.3590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7999 0.7412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1240 -0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7860 -0.4055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7860 0.3590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1240 0.7412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1240 -1.5562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4477 0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1225 0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7973 0.7411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7973 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1225 1.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4477 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7999 -1.5504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5391 0.8104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5752 -0.8172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4719 1.9097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6898 0.7620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0844 -0.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2126 0.3019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3713 0.3110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9826 0.9224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8732 0.6027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3668 0.3713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0282 -0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4848 -0.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0631 -1.8048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4101 -1.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6185 -1.7371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8641 -1.1840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0538 -0.9575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8025 -0.5250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5800 -1.0645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2437 -1.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8125 -1.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3969 -0.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3619 -0.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4719 -1.1786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3038 1.1278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2724 1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8177 1.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
2 20 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
31 32 1 0 0 0 0
35 30 1 0 0 0 0
25 19 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
37 40 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
27 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 40 41 42
M SBL 1 1 46
M SMT 1 ^ COOH
M SBV 1 46 0.8168 -0.3584
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 43 44 45
M SBL 2 1 49
M SMT 2 ^ COOH
M SBV 2 49 0.4306 -0.5251
S SKP 5
ID FL3FEAGS0030
FORMULA C27H26O18
EXACTMASS 638.111914028
AVERAGEMASS 638.4845399999999
SMILES c(c1)(ccc(C(O2)=CC(c(c(O)5)c2cc(c(O)5)OC(C3OC(O4)C(O)C(C(C4C(O)=O)O)O)OC(C(C3O)O)C(O)=O)=O)c1)O
M END