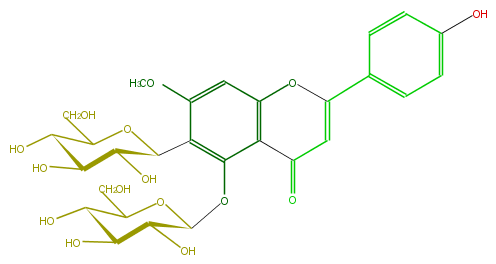
Mol:FL3FCADS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1178 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1178 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1178 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1178 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4343 -0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4343 -0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2492 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2492 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2492 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2492 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4343 1.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4343 1.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9327 -0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9327 -0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6162 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6162 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6162 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6162 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9327 1.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9327 1.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9327 -1.3538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9327 -1.3538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4343 -1.3674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4343 -1.3674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4514 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4514 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1717 0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1717 0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8920 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8920 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8920 1.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8920 1.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1717 2.3741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1717 2.3741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4514 1.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4514 1.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6118 2.3738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6118 2.3738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8973 -0.0494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8973 -0.0494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2638 -0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2638 -0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6303 -0.3789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6303 -0.3789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7686 -0.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7686 -0.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3768 0.0773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3768 0.0773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0611 -0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0611 -0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5085 -0.3336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5085 -0.3336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0517 -0.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0517 -0.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0118 -0.9231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0118 -0.9231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2307 -1.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2307 -1.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5972 -2.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5972 -2.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9638 -1.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9638 -1.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1021 -1.9394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1021 -1.9394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7103 -1.3820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7103 -1.3820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3945 -1.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3945 -1.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9120 -1.7683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9120 -1.7683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3852 -2.1972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3852 -2.1972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2316 -2.3741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2316 -2.3741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6930 0.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6930 0.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6118 0.7922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6118 0.7922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7837 0.9898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7837 0.9898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3980 2.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3980 2.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9831 -1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9831 -1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9591 -1.8040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9591 -1.8040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 2 1 0 0 0 0 | + | 23 2 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 12 1 0 0 0 0 | + | 32 12 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 1 40 1 0 0 0 0 | + | 1 40 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 43 0.6319 -0.6159 | + | M SBV 1 43 0.6319 -0.6159 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 45 0.6659 -0.3845 | + | M SBV 2 45 0.6659 -0.3845 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 42 43 | + | M SAL 3 2 42 43 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 47 0.5886 -0.5859 | + | M SBV 3 47 0.5886 -0.5859 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCADS0005 | + | ID FL3FCADS0005 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES C(c12)(=O)C=C(c(c5)ccc(O)c5)Oc1cc(OC)c(C(C4O)OC(CO)C(C(O)4)O)c2OC(O3)C(O)C(C(C(CO)3)O)O | + | SMILES C(c12)(=O)C=C(c(c5)ccc(O)c5)Oc1cc(OC)c(C(C4O)OC(CO)C(C(O)4)O)c2OC(O3)C(O)C(C(C(CO)3)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.1178 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1178 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4343 -0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2492 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2492 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4343 1.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9327 -0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6162 -0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6162 0.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9327 1.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9327 -1.3538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4343 -1.3674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4514 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1717 0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8920 1.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8920 1.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1717 2.3741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4514 1.9582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6118 2.3738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8973 -0.0494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2638 -0.7589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6303 -0.3789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7686 -0.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3768 0.0773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0611 -0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5085 -0.3336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0517 -0.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0118 -0.9231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2307 -1.5086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5972 -2.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9638 -1.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1021 -1.9394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7103 -1.3820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3945 -1.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9120 -1.7683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3852 -2.1972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2316 -2.3741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6930 0.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6118 0.7922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7837 0.9898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3980 2.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9831 -1.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9591 -1.8040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 2 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 12 1 0 0 0 0
38 39 1 0 0 0 0
25 38 1 0 0 0 0
40 41 1 0 0 0 0
1 40 1 0 0 0 0
42 43 1 0 0 0 0
34 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 ^ CH2OH
M SBV 1 43 0.6319 -0.6159
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 ^ OCH3
M SBV 2 45 0.6659 -0.3845
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 42 43
M SBL 3 1 47
M SMT 3 ^ CH2OH
M SBV 3 47 0.5886 -0.5859
S SKP 5
ID FL3FCADS0005
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES C(c12)(=O)C=C(c(c5)ccc(O)c5)Oc1cc(OC)c(C(C4O)OC(CO)C(C(O)4)O)c2OC(O3)C(O)C(C(C(CO)3)O)O
M END