Mol:FL3FCADS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8324 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8324 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8324 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8324 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2761 -1.9332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2761 -1.9332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7198 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7198 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7198 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7198 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2761 -0.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2761 -0.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1635 -1.9332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1635 -1.9332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3928 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3928 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3928 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3928 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1635 -0.6485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1635 -0.6485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1635 -2.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1635 -2.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2761 -2.5754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2761 -2.5754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0727 -0.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0727 -0.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6589 -0.8840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6589 -0.8840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2451 -0.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2451 -0.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2451 0.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2451 0.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6589 0.4698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6589 0.4698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0727 0.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0727 0.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9567 0.6184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9567 0.6184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3858 -1.8566 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.3858 -1.8566 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.8702 -2.4341 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.8702 -2.4341 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3545 -2.1247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3545 -2.1247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6533 -2.2072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6533 -2.2072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.1483 -1.7534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1483 -1.7534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7052 -2.0216 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.7052 -2.0216 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.8976 -2.1521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8976 -2.1521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5125 -2.5166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5125 -2.5166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0452 -2.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0452 -2.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6745 0.2884 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.6745 0.2884 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.3156 0.5997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3156 0.5997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2895 1.1586 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.2895 1.1586 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.5627 1.6409 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.5627 1.6409 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.9626 1.4291 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.9626 1.4291 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.0156 0.8468 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.0156 0.8468 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.7907 1.1654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7907 1.1654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2127 2.3624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2127 2.3624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4566 0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4566 0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5627 2.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5627 2.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7727 -1.5027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7727 -1.5027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9160 -0.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9160 -0.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1896 -0.3509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1896 -0.3509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6895 0.5152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6895 0.5152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 19 29 1 0 0 0 0 | + | 19 29 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 39 40 | + | M SAL 2 2 39 40 |
| − | M SBL 2 1 43 | + | M SBL 2 1 43 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 43 -4.0581 -1.6353 | + | M SVB 2 43 -4.0581 -1.6353 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 45 -2.1896 -0.3509 | + | M SVB 1 45 -2.1896 -0.3509 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FCADS0002 | + | ID FL3FCADS0002 |
| − | KNApSAcK_ID C00006267 | + | KNApSAcK_ID C00006267 |
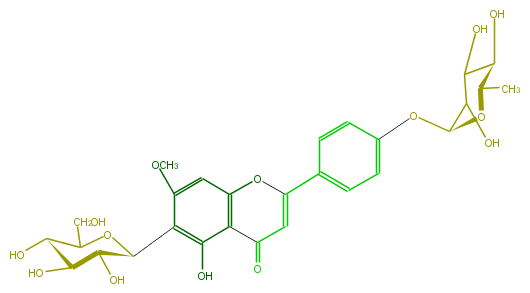
| − | NAME Flavocummelin;Swertisin 4'-O-rhamnoside | + | NAME Flavocummelin;Swertisin 4'-O-rhamnoside |
| − | CAS_RN 68027-86-1 | + | CAS_RN 68027-86-1 |
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES O[C@H]([C@@H]1c(c(OC)2)c(c(C5=O)c(OC(=C5)c(c3)ccc(O[C@H](O4)C(O)C([C@@H](O)[C@H]4C)O)c3)c2)O)[C@H]([C@H](C(O1)CO)O)O | + | SMILES O[C@H]([C@@H]1c(c(OC)2)c(c(C5=O)c(OC(=C5)c(c3)ccc(O[C@H](O4)C(O)C([C@@H](O)[C@H]4C)O)c3)c2)O)[C@H]([C@H](C(O1)CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.8324 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8324 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2761 -1.9332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7198 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7198 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2761 -0.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1635 -1.9332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3928 -1.6121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3928 -0.9697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1635 -0.6485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1635 -2.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2761 -2.5754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0727 -0.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6589 -0.8840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2451 -0.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2451 0.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6589 0.4698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0727 0.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9567 0.6184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3858 -1.8566 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.8702 -2.4341 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3545 -2.1247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6533 -2.2072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.1483 -1.7534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7052 -2.0216 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.8976 -2.1521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5125 -2.5166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0452 -2.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6745 0.2884 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.3156 0.5997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2895 1.1586 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.5627 1.6409 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.9626 1.4291 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.0156 0.8468 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.7907 1.1654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2127 2.3624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4566 0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5627 2.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7727 -1.5027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9160 -0.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1896 -0.3509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6895 0.5152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
2 23 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
31 35 1 0 0 0 0
33 36 1 0 0 0 0
19 29 1 0 0 0 0
34 37 1 0 0 0 0
31 30 1 1 0 0 0
32 38 1 0 0 0 0
25 39 1 0 0 0 0
39 40 1 0 0 0 0
1 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 39 40
M SBL 2 1 43
M SMT 2 CH2OH
M SVB 2 43 -4.0581 -1.6353
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 OCH3
M SVB 1 45 -2.1896 -0.3509
S SKP 8
ID FL3FCADS0002
KNApSAcK_ID C00006267
NAME Flavocummelin;Swertisin 4'-O-rhamnoside
CAS_RN 68027-86-1
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES O[C@H]([C@@H]1c(c(OC)2)c(c(C5=O)c(OC(=C5)c(c3)ccc(O[C@H](O4)C(O)C([C@@H](O)[C@H]4C)O)c3)c2)O)[C@H]([C@H](C(O1)CO)O)O
M END