Mol:FL3FCACS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3208 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3208 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3208 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3208 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6060 -0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6060 -0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1087 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1087 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1087 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1087 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6060 1.3572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6060 1.3572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8235 -0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8235 -0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5382 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5382 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5382 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5382 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8235 1.3572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8235 1.3572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8235 -0.9369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8235 -0.9369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6060 -1.1184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6060 -1.1184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3323 1.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3323 1.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0854 0.9488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0854 0.9488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8386 1.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8386 1.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8386 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8386 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0854 2.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0854 2.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3323 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3323 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5914 2.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5914 2.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6689 0.1464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6689 0.1464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2241 -0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2241 -0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5835 -0.1918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5835 -0.1918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9158 -0.1991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9158 -0.1991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4145 0.2642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4145 0.2642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0689 0.0292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0689 0.0292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3633 0.0300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3633 0.0300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0432 -0.4152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0432 -0.4152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9057 -0.7062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9057 -0.7062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0414 -1.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0414 -1.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3759 -1.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3759 -1.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7327 -1.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7327 -1.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0856 -1.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0856 -1.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7511 -1.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7511 -1.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3943 -1.5988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3943 -1.5988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6598 -1.4517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6598 -1.4517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9404 -1.8100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9404 -1.8100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1560 -2.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1560 -2.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0644 -2.6882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0644 -2.6882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6307 0.5755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6307 0.5755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5914 1.0236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5914 1.0236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8609 1.2845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8609 1.2845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5033 2.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5033 2.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 23 2 1 0 0 0 0 | + | 23 2 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 44 0.5618 -0.5463 | + | M SBV 1 44 0.5618 -0.5463 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 46 0.5401 -0.3398 | + | M SBV 2 46 0.5401 -0.3398 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCACS0016 | + | ID FL3FCACS0016 |
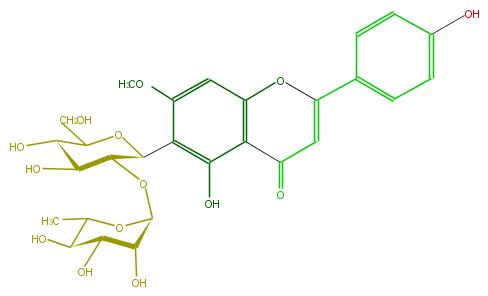
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES c(c5)c(ccc5O)C(O4)=CC(c(c34)c(c(c(c3)OC)C(C1OC(O2)C(O)C(O)C(C(C)2)O)OC(C(O)C1O)CO)O)=O | + | SMILES c(c5)c(ccc5O)C(O4)=CC(c(c34)c(c(c(c3)OC)C(C1OC(O2)C(O)C(O)C(C(C)2)O)OC(C(O)C1O)CO)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.3208 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3208 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6060 -0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1087 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1087 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6060 1.3572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8235 -0.2934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5382 0.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5382 0.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8235 1.3572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8235 -0.9369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6060 -1.1184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3323 1.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0854 0.9488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8386 1.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8386 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0854 2.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3323 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5914 2.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6689 0.1464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2241 -0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5835 -0.1918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9158 -0.1991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4145 0.2642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0689 0.0292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3633 0.0300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0432 -0.4152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9057 -0.7062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0414 -1.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3759 -1.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7327 -1.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0856 -1.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7511 -1.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3943 -1.5988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6598 -1.4517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9404 -1.8100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1560 -2.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0644 -2.6882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6307 0.5755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5914 1.0236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8609 1.2845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5033 2.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 28 1 0 0 0 0
23 2 1 0 0 0 0
39 40 1 0 0 0 0
25 39 1 0 0 0 0
41 42 1 0 0 0 0
1 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ CH2OH
M SBV 1 44 0.5618 -0.5463
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 ^ OCH3
M SBV 2 46 0.5401 -0.3398
S SKP 5
ID FL3FCACS0016
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES c(c5)c(ccc5O)C(O4)=CC(c(c34)c(c(c(c3)OC)C(C1OC(O2)C(O)C(O)C(C(C)2)O)OC(C(O)C1O)CO)O)=O
M END