Mol:FL3FAHGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 51 0 0 0 0 0 0 0 0999 V2000 | + | 47 51 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9047 -0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9047 -0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9047 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9047 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2132 -1.9751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2132 -1.9751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5220 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5220 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5220 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5220 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2132 -0.3784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2132 -0.3784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8305 -1.9751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8305 -1.9751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1391 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1391 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1391 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1391 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8305 -0.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8305 -0.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8305 -2.7398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8305 -2.7398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5520 -0.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5520 -0.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2567 -0.7853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2567 -0.7853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9613 -0.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9613 -0.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9613 0.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9613 0.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2567 0.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2567 0.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5520 0.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5520 0.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2132 -2.7717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2132 -2.7717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7006 0.8621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7006 0.8621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6659 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6659 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5836 -0.3043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5836 -0.3043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0462 2.2655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0462 2.2655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4275 1.3609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4275 1.3609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7133 0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7133 0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2938 0.0789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2938 0.0789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0746 0.8965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0746 0.8965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7816 1.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7816 1.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6858 2.6687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6858 2.6687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7591 2.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7591 2.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7445 -0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7445 -0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8671 -0.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8671 -0.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0953 -1.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0953 -1.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9599 -1.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9599 -1.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5840 -2.1593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5840 -2.1593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3560 -1.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3560 -1.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4913 -1.4973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4913 -1.4973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2635 -0.6151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2635 -0.6151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2894 -1.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2894 -1.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7591 -2.1660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7591 -2.1660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3092 -2.5322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3092 -2.5322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7099 -3.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7099 -3.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4878 -3.0064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4878 -3.0064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2567 1.6757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2567 1.6757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8520 3.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8520 3.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4865 2.3532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4865 2.3532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9935 3.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9935 3.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4090 1.9525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4090 1.9525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 21 1 1 0 0 0 0 | + | 21 1 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 20 1 0 0 0 0 | + | 32 20 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 16 43 1 0 0 0 0 | + | 16 43 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 27 45 1 0 0 0 0 | + | 27 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 40 41 42 | + | M SAL 1 3 40 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SBV 1 46 -0.7252 0.3729 | + | M SBV 1 46 -0.7252 0.3729 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 48 0.0000 -0.8337 | + | M SBV 2 48 0.0000 -0.8337 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 3 45 46 47 | + | M SAL 3 3 45 46 47 |
| − | M SBL 3 1 51 | + | M SBL 3 1 51 |
| − | M SMT 3 COOH | + | M SMT 3 COOH |
| − | M SBV 3 51 -0.2951 -0.8580 | + | M SBV 3 51 -0.2951 -0.8580 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAHGS0002 | + | ID FL3FAHGS0002 |
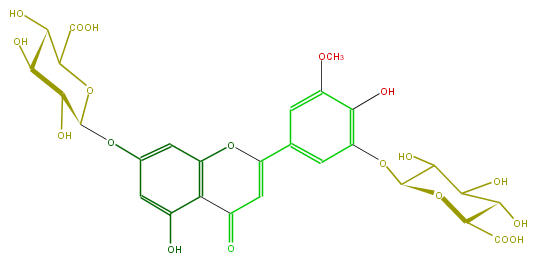
| − | FORMULA C28H28O19 | + | FORMULA C28H28O19 |
| − | EXACTMASS 668.122478714 | + | EXACTMASS 668.122478714 |
| − | AVERAGEMASS 668.51052 | + | AVERAGEMASS 668.51052 |
| − | SMILES OC(C(C(O)=O)5)C(C(C(O5)Oc(c1)cc(c(C4=O)c1OC(=C4)c(c2)cc(OC)c(c2OC(C3O)OC(C(O)=O)C(O)C3O)O)O)O)O | + | SMILES OC(C(C(O)=O)5)C(C(C(O5)Oc(c1)cc(c(C4=O)c1OC(=C4)c(c2)cc(OC)c(c2OC(C3O)OC(C(O)=O)C(O)C3O)O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 51 0 0 0 0 0 0 0 0999 V2000
-2.9047 -0.7144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9047 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2132 -1.9751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5220 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5220 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2132 -0.3784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8305 -1.9751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1391 -1.5759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1391 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8305 -0.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8305 -2.7398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5520 -0.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2567 -0.7853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9613 -0.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9613 0.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2567 0.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5520 0.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2132 -2.7717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7006 0.8621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6659 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5836 -0.3043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0462 2.2655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4275 1.3609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7133 0.7877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2938 0.0789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0746 0.8965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7816 1.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6858 2.6687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7591 2.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7445 -0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8671 -0.8541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0953 -1.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9599 -1.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5840 -2.1593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3560 -1.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4913 -1.4973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2635 -0.6151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2894 -1.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7591 -2.1660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3092 -2.5322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7099 -3.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4878 -3.0064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2567 1.6757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8520 3.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4865 2.3532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9935 3.3917 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4090 1.9525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
14 20 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 21 1 0 0 0 0
21 1 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 20 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
34 40 1 0 0 0 0
43 44 1 0 0 0 0
16 43 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
27 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 40 41 42
M SBL 1 1 46
M SMT 1 COOH
M SBV 1 46 -0.7252 0.3729
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 48
M SMT 2 OCH3
M SBV 2 48 0.0000 -0.8337
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 3 45 46 47
M SBL 3 1 51
M SMT 3 COOH
M SBV 3 51 -0.2951 -0.8580
S SKP 5
ID FL3FAHGS0002
FORMULA C28H28O19
EXACTMASS 668.122478714
AVERAGEMASS 668.51052
SMILES OC(C(C(O)=O)5)C(C(C(O5)Oc(c1)cc(c(C4=O)c1OC(=C4)c(c2)cc(OC)c(c2OC(C3O)OC(C(O)=O)C(O)C3O)O)O)O)O
M END