Mol:FL3FACGS0073
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.3206 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3206 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3206 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3206 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0351 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0351 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7496 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7496 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7496 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7496 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0351 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0351 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4640 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4640 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1784 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1784 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1784 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1784 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4640 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4640 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8929 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8929 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6074 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6074 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3218 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3218 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3218 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3218 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6074 1.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6074 1.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8929 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8929 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4640 -1.8397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4640 -1.8397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3990 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3990 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0351 -1.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0351 -1.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9446 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9446 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0275 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0275 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4487 0.3203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4487 0.3203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6019 -0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6019 -0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9279 -0.0143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9279 -0.0143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1054 0.0521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1054 0.0521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9520 0.8630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9520 0.8630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6261 0.3867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6261 0.3867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7494 0.9340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7494 0.9340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1224 -0.4906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1224 -0.4906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1052 -0.6761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1052 -0.6761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4894 1.3065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4894 1.3065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0768 0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0768 0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2784 0.8010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2784 0.8010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4850 0.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4850 0.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8975 1.2889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8975 1.2889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6959 1.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6959 1.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1213 1.5734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1213 1.5734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8641 1.5734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8641 1.5734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0275 0.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0275 0.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6170 0.5918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6170 0.5918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9468 0.2267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9468 0.2267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 28 1 0 0 0 0 | + | 34 28 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
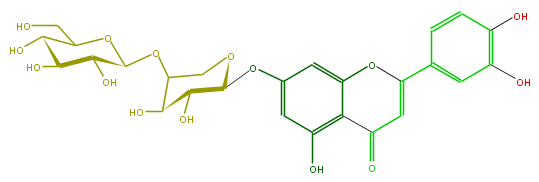
| − | ID FL3FACGS0073 | + | ID FL3FACGS0073 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES c(c1)c(c(O)cc1C(O5)=CC(=O)c(c45)c(O)cc(c4)OC(C(O)3)OCC(C3O)OC(O2)C(C(C(C2CO)O)O)O)O | + | SMILES c(c1)c(c(O)cc1C(O5)=CC(=O)c(c45)c(O)cc(c4)OC(C(O)3)OCC(C3O)OC(O2)C(C(C(C2CO)O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
0.3206 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3206 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0351 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7496 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7496 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0351 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4640 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1784 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1784 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4640 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8929 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6074 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3218 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3218 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6074 1.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8929 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4640 -1.8397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3990 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0351 -1.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9446 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0275 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4487 0.3203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6019 -0.4906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9279 -0.0143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1054 0.0521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9520 0.8630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6261 0.3867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7494 0.9340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1224 -0.4906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1052 -0.6761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4894 1.3065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0768 0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2784 0.8010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4850 0.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8975 1.2889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6959 1.0798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1213 1.5734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8641 1.5734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0275 0.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6170 0.5918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9468 0.2267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
14 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 18 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
31 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 28 1 0 0 0 0
S SKP 5
ID FL3FACGS0073
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES c(c1)c(c(O)cc1C(O5)=CC(=O)c(c45)c(O)cc(c4)OC(C(O)3)OCC(C3O)OC(O2)C(C(C(C2CO)O)O)O)O
M END