Mol:FL3FACGS0040
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.4110 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4110 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4110 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4110 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9599 -1.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9599 -1.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5088 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5088 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5088 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5088 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9599 -0.4363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9599 -0.4363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0578 -1.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0578 -1.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6067 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6067 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6067 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6067 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0578 -0.4363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0578 -0.4363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0578 -1.8841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0578 -1.8841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1558 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1558 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6961 -0.7018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6961 -0.7018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2364 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2364 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2364 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2364 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6961 0.3599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6961 0.3599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1558 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1558 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9304 -0.3968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9304 -0.3968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9599 -1.9977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9599 -1.9977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3580 0.4376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3580 0.4376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6876 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6876 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3000 -0.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3000 -0.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0454 0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0454 0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7954 -0.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7954 -0.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1831 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1831 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4376 0.4411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4376 0.4411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9677 0.4611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9677 0.4611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7446 0.9727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7446 0.9727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7425 0.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7425 0.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4456 1.3261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4456 1.3261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9299 0.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9299 0.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1874 0.9342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1874 0.9342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4710 0.9419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4710 0.9419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9916 1.4627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9916 1.4627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7501 1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7501 1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0221 0.9932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0221 0.9932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7337 0.6063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7337 0.6063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7620 0.2200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7620 0.2200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8828 1.5301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8828 1.5301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3076 0.0093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3076 0.0093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0221 -0.4032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0221 -0.4032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0456 1.9977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0456 1.9977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3311 1.5852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3311 1.5852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 39 16 1 0 0 0 0 | + | 39 16 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -8.8717 4.0426 | + | M SBV 1 44 -8.8717 4.0426 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 46 -9.6794 4.8233 | + | M SBV 2 46 -9.6794 4.8233 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACGS0040 | + | ID FL3FACGS0040 |
| − | KNApSAcK_ID C00004300 | + | KNApSAcK_ID C00004300 |
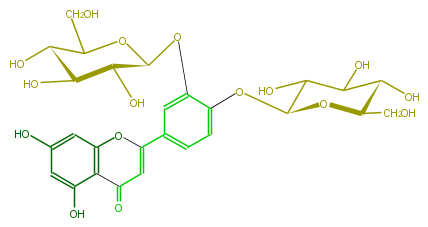
| − | NAME Luteolin 3',4'-diglucoside | + | NAME Luteolin 3',4'-diglucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES Oc(c1)cc(c(C(=O)5)c1OC(=C5)c(c3)ccc(c3OC(O4)C(C(C(O)C4CO)O)O)OC(O2)C(C(C(C(CO)2)O)O)O)O | + | SMILES Oc(c1)cc(c(C(=O)5)c1OC(=C5)c(c3)ccc(c3OC(O4)C(C(C(O)C4CO)O)O)OC(O2)C(C(C(C(CO)2)O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-3.4110 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4110 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9599 -1.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5088 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5088 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9599 -0.4363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0578 -1.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6067 -1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6067 -0.6967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0578 -0.4363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0578 -1.8841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1558 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6961 -0.7018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2364 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2364 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6961 0.3599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1558 0.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9304 -0.3968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9599 -1.9977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3580 0.4376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6876 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3000 -0.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0454 0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7954 -0.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1831 0.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4376 0.4411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9677 0.4611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7446 0.9727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7425 0.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4456 1.3261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9299 0.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1874 0.9342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4710 0.9419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9916 1.4627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7501 1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0221 0.9932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7337 0.6063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7620 0.2200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8828 1.5301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3076 0.0093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0221 -0.4032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0456 1.9977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3311 1.5852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
39 16 1 0 0 0 0
24 40 1 0 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -8.8717 4.0426
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 ^CH2OH
M SBV 2 46 -9.6794 4.8233
S SKP 8
ID FL3FACGS0040
KNApSAcK_ID C00004300
NAME Luteolin 3',4'-diglucoside
CAS_RN -
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES Oc(c1)cc(c(C(=O)5)c1OC(=C5)c(c3)ccc(c3OC(O4)C(C(C(O)C4CO)O)O)OC(O2)C(C(C(C(CO)2)O)O)O)O
M END