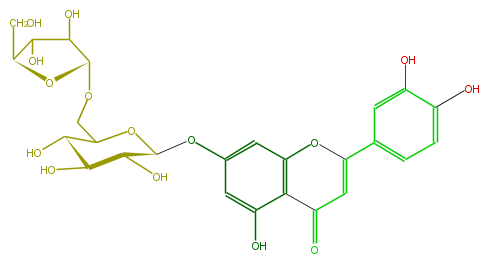
Mol:FL3FACGS0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1451 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1451 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1451 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1451 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7760 -1.7359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7760 -1.7359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4067 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4067 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4067 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4067 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7760 -0.2790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7760 -0.2790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0376 -1.7359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0376 -1.7359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6685 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6685 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6685 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6685 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0376 -0.2790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0376 -0.2790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0315 -2.5404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0315 -2.5404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2990 -0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2990 -0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9420 -0.6504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9420 -0.6504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5849 -0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5849 -0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5849 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5849 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9420 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9420 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2990 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2990 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5815 -0.2238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5815 -0.2238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7760 -2.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7760 -2.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2937 0.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2937 0.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9434 1.4802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9434 1.4802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2646 -0.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2646 -0.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7598 -0.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7598 -0.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0329 -0.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0329 -0.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3315 -0.5358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3315 -0.5358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8411 -0.0260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8411 -0.0260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5837 -0.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5837 -0.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8290 -0.4855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8290 -0.4855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5467 -0.8644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5467 -0.8644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3056 -0.9965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3056 -0.9965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0042 0.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0042 0.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1670 1.8850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1670 1.8850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9555 1.8850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9555 1.8850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3582 1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3582 1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5708 0.9966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5708 0.9966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7429 1.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7429 1.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9435 1.4617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9435 1.4617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1659 2.4576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1659 2.4576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7450 0.7150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7450 0.7150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3569 2.2562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3569 2.2562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2937 2.5404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2937 2.5404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 39 31 1 0 0 0 0 | + | 39 31 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 45 -0.0013 -0.7952 | + | M SBV 1 45 -0.0013 -0.7952 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACGS0019 | + | ID FL3FACGS0019 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES c(c3)(c(c(O)cc(OC(C(O)4)OC(COC(C(O)5)OC(CO)C5O)C(O)C4O)3)2)OC(=CC(=O)2)c(c1)cc(c(c1)O)O | + | SMILES c(c3)(c(c(O)cc(OC(C(O)4)OC(COC(C(O)5)OC(CO)C5O)C(O)C4O)3)2)OC(=CC(=O)2)c(c1)cc(c(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
0.1451 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1451 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7760 -1.7359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4067 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4067 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7760 -0.2790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0376 -1.7359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6685 -1.3716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6685 -0.6432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0376 -0.2790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0315 -2.5404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2990 -0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9420 -0.6504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5849 -0.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5849 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9420 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2990 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5815 -0.2238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7760 -2.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2937 0.8593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9434 1.4802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2646 -0.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7598 -0.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0329 -0.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3315 -0.5358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8411 -0.0260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5837 -0.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8290 -0.4855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5467 -0.8644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3056 -0.9965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0042 0.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1670 1.8850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9555 1.8850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3582 1.4610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5708 0.9966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7429 1.4206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9435 1.4617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1659 2.4576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7450 0.7150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3569 2.2562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2937 2.5404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 18 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 32 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
36 39 1 0 0 0 0
39 31 1 0 0 0 0
40 41 1 0 0 0 0
34 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 CH2OH
M SBV 1 45 -0.0013 -0.7952
S SKP 5
ID FL3FACGS0019
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES c(c3)(c(c(O)cc(OC(C(O)4)OC(COC(C(O)5)OC(CO)C5O)C(O)C4O)3)2)OC(=CC(=O)2)c(c1)cc(c(c1)O)O
M END