Mol:FL3FACDS0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4908 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4908 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4908 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4908 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7764 -3.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7764 -3.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0619 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0619 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0619 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0619 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7764 -1.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7764 -1.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6525 -3.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6525 -3.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3670 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3670 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3670 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3670 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6525 -1.7124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6525 -1.7124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6525 -4.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6525 -4.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2051 -1.7125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2051 -1.7125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7764 -4.1870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7764 -4.1870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2402 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2402 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9931 -2.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9931 -2.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7459 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7459 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7459 -0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7459 -0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9931 -0.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9931 -0.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2402 -0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2402 -0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4984 -0.2762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4984 -0.2762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1619 3.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1619 3.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3841 2.9500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3841 2.9500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7140 2.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7140 2.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7229 1.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7229 1.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3179 1.8778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3179 1.8778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9262 2.6201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9262 2.6201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7272 4.1870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7272 4.1870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3408 3.6816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3408 3.6816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2694 1.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2694 1.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7412 1.5170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7412 1.5170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4158 1.1275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4158 1.1275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2018 1.8765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2018 1.8765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4158 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4158 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7412 3.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7412 3.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9553 2.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9553 2.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0286 3.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0286 3.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4378 3.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4378 3.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3498 2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3498 2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1522 1.5073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1522 1.5073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1498 0.5350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1498 0.5350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9636 -2.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9636 -2.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4869 -3.5789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4869 -3.5789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8004 -3.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8004 -3.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1381 -3.3047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1381 -3.3047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6194 -2.8234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6194 -2.8234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2199 -3.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2199 -3.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4984 -3.2083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4984 -3.2083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0636 -3.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0636 -3.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1788 -3.8498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1788 -3.8498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4923 3.2788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4923 3.2788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4099 2.7490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4099 2.7490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7289 -2.5897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7289 -2.5897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7055 -2.8639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7055 -2.8639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 9 14 1 0 0 0 0 | + | 9 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 31 29 1 0 0 0 0 | + | 31 29 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 40 18 1 0 0 0 0 | + | 40 18 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 2 1 0 0 0 0 | + | 44 2 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 26 50 1 0 0 0 0 | + | 26 50 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 56 -0.5660 -0.6587 | + | M SBV 1 56 -0.5660 -0.6587 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 58 0.5090 -0.5507 | + | M SBV 2 58 0.5090 -0.5507 |
| − | S SKP 5 | + | S SKP 5 |
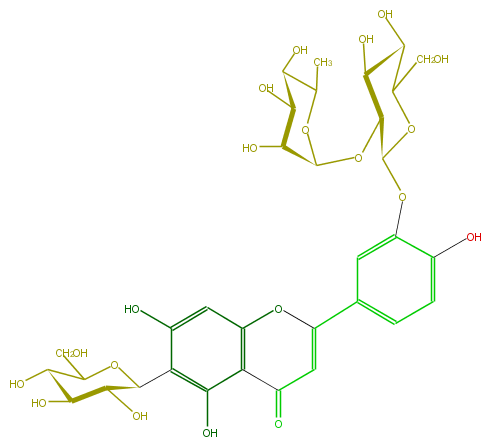
| − | ID FL3FACDS0014 | + | ID FL3FACDS0014 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES OC(C(CO)1)C(O)C(C(c(c6O)c(c(c2c6)C(=O)C=C(c(c3)ccc(O)c3OC(C4OC(C5O)OC(C)C(C5O)O)OC(C(O)C4O)CO)O2)O)O1)O | + | SMILES OC(C(CO)1)C(O)C(C(c(c6O)c(c(c2c6)C(=O)C=C(c(c3)ccc(O)c3OC(C4OC(C5O)OC(C)C(C5O)O)OC(C(O)C4O)CO)O2)O)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-1.4908 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4908 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7764 -3.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0619 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0619 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7764 -1.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6525 -3.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3670 -2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3670 -2.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6525 -1.7124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6525 -4.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2051 -1.7125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7764 -4.1870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2402 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9931 -2.0147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7459 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7459 -0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9931 -0.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2402 -0.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4984 -0.2762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1619 3.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3841 2.9500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7140 2.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7229 1.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3179 1.8778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9262 2.6201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7272 4.1870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3408 3.6816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2694 1.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7412 1.5170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4158 1.1275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2018 1.8765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4158 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7412 3.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9553 2.2705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0286 3.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4378 3.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3498 2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1522 1.5073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1498 0.5350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9636 -2.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4869 -3.5789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8004 -3.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1381 -3.3047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6194 -2.8234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2199 -3.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4984 -3.2083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0636 -3.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1788 -3.8498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4923 3.2788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4099 2.7490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7289 -2.5897 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7055 -2.8639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
9 14 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
35 38 1 0 0 0 0
30 39 1 0 0 0 0
31 29 1 0 0 0 0
24 40 1 0 0 0 0
40 18 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 2 1 0 0 0 0
50 51 1 0 0 0 0
26 50 1 0 0 0 0
52 53 1 0 0 0 0
46 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 56
M SMT 1 CH2OH
M SBV 1 56 -0.5660 -0.6587
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 58
M SMT 2 ^CH2OH
M SBV 2 58 0.5090 -0.5507
S SKP 5
ID FL3FACDS0014
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES OC(C(CO)1)C(O)C(C(c(c6O)c(c(c2c6)C(=O)C=C(c(c3)ccc(O)c3OC(C4OC(C5O)OC(C)C(C5O)O)OC(C(O)C4O)CO)O2)O)O1)O
M END