Mol:FL3FACDS0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5439 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5439 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5439 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5439 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9876 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9876 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4313 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4313 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4313 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4313 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9876 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9876 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8750 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8750 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3187 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3187 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3187 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3187 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8750 -0.7513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8750 -0.7513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8750 -2.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8750 -2.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1000 -0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1000 -0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7628 1.7270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7628 1.7270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3684 1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3684 1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1115 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1115 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1046 -0.0299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1046 -0.0299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6413 0.4333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6413 0.4333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9463 1.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9463 1.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0177 2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0177 2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4032 1.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4032 1.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7470 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7470 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9876 -2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9876 -2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6389 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6389 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0526 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0526 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5336 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5336 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5336 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5336 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0526 0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0526 0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6389 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6389 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1194 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1194 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1729 -0.5679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1729 -0.5679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8721 -1.0890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8721 -1.0890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4506 -0.9236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4506 -0.9236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0326 -1.0890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0326 -1.0890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3335 -0.5679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3335 -0.5679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7549 -0.7332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7549 -0.7332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6142 -0.7176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6142 -0.7176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9931 -0.3206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9931 -0.3206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7675 -0.8185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7675 -0.8185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2088 -0.9112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2088 -0.9112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5097 1.2633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5097 1.2633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7953 0.8508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7953 0.8508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3855 -1.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3855 -1.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1000 -1.4747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1000 -1.4747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 9 23 1 0 0 0 0 | + | 9 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 18 40 1 0 0 0 0 | + | 18 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -8.1563 5.7796 | + | M SBV 1 44 -8.1563 5.7796 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 -8.2399 5.5543 | + | M SBV 2 46 -8.2399 5.5543 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACDS0009 | + | ID FL3FACDS0009 |
| − | KNApSAcK_ID C00006253 | + | KNApSAcK_ID C00006253 |
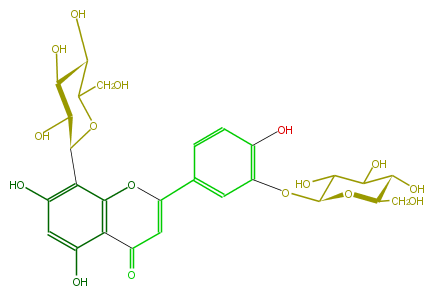
| − | NAME Orientin 3'-O-glucoside | + | NAME Orientin 3'-O-glucoside |
| − | CAS_RN 85708-47-0 | + | CAS_RN 85708-47-0 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES c(c(O)4)(c(c(C(C(O)5)OC(C(O)C(O)5)CO)c(c4)O)3)C(C=C(O3)c(c1)ccc(c1OC(O2)C(C(C(O)C(CO)2)O)O)O)=O | + | SMILES c(c(O)4)(c(c(C(C(O)5)OC(C(O)C(O)5)CO)c(c4)O)3)C(C=C(O3)c(c1)ccc(c1OC(O2)C(C(C(O)C(CO)2)O)O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-3.5439 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5439 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9876 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4313 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4313 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9876 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8750 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3187 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3187 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8750 -0.7513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8750 -2.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1000 -0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7628 1.7270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3684 1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1115 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1046 -0.0299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6413 0.4333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9463 1.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0177 2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4032 1.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7470 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9876 -2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6389 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0526 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5336 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5336 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0526 0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6389 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1194 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1729 -0.5679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8721 -1.0890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4506 -0.9236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0326 -1.0890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3335 -0.5679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7549 -0.7332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6142 -0.7176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9931 -0.3206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7675 -0.8185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2088 -0.9112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5097 1.2633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7953 0.8508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3855 -1.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1000 -1.4747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
9 23 1 0 0 0 0
26 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
31 39 1 0 0 0 0
25 39 1 0 0 0 0
18 40 1 0 0 0 0
40 41 1 0 0 0 0
33 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -8.1563 5.7796
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 -8.2399 5.5543
S SKP 8
ID FL3FACDS0009
KNApSAcK_ID C00006253
NAME Orientin 3'-O-glucoside
CAS_RN 85708-47-0
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES c(c(O)4)(c(c(C(C(O)5)OC(C(O)C(O)5)CO)c(c4)O)3)C(C=C(O3)c(c1)ccc(c1OC(O2)C(C(C(O)C(CO)2)O)O)O)=O
M END