Mol:FL3FACCS0058
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.8267 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8267 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1122 0.5662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1122 0.5662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3978 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3978 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3978 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3978 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1122 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1122 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8267 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8267 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6833 0.5662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6833 0.5662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9688 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9688 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9688 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9688 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6833 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6833 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1122 -1.6559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1122 -1.6559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6281 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6281 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3425 0.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3425 0.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0570 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0570 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0570 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0570 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3425 1.8538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3425 1.8538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6281 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6281 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7405 1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7405 1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6833 -1.8538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6833 -1.8538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2783 0.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2783 0.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7185 0.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7185 0.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8845 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8845 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4718 -1.0226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4718 -1.0226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6735 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6735 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1200 -1.0403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1200 -1.0403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2926 -0.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2926 -0.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0910 -0.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0910 -0.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2480 0.0512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2480 0.0512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3677 -0.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3677 -0.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9205 -0.7635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9205 -0.7635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8918 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8918 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7949 0.4082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7949 0.4082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2069 1.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2069 1.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7949 1.8352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7949 1.8352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0307 1.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0307 1.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4427 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4427 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2666 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2666 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6791 1.1226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6791 1.1226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5041 1.1226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5041 1.1226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9166 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9166 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5041 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5041 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6791 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6791 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7405 0.4082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7405 0.4082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9160 1.8361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9160 1.8361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 14 21 1 0 0 0 0 | + | 14 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 25 9 1 0 0 0 0 | + | 25 9 1 0 0 0 0 |
| − | 31 24 1 0 0 0 0 | + | 31 24 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 39 44 1 0 0 0 0 | + | 39 44 1 0 0 0 0 |
| − | 32 28 1 0 0 0 0 | + | 32 28 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACCS0058 | + | ID FL3FACCS0058 |
| − | KNApSAcK_ID C00014100 | + | KNApSAcK_ID C00014100 |
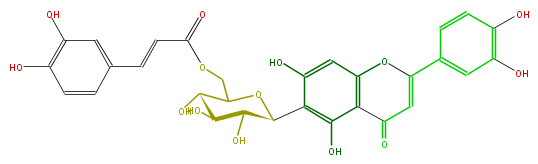
| − | NAME Isoorientin 6''-O-caffeate | + | NAME Isoorientin 6''-O-caffeate |
| − | CAS_RN 172377-88-7 | + | CAS_RN 172377-88-7 |
| − | FORMULA C30H26O14 | + | FORMULA C30H26O14 |
| − | EXACTMASS 610.13225554 | + | EXACTMASS 610.13225554 |
| − | AVERAGEMASS 610.51904 | + | AVERAGEMASS 610.51904 |
| − | SMILES c(c5)c(cc(O)c(O)5)C(=C4)Oc(c3)c(C(=O)4)c(c(c3O)C(C1O)OC(COC(=O)C=Cc(c2)ccc(O)c2O)C(C1O)O)O | + | SMILES c(c5)c(cc(O)c(O)5)C(=C4)Oc(c3)c(C(=O)4)c(c(c3O)C(C1O)OC(COC(=O)C=Cc(c2)ccc(O)c2O)C(C1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
3.8267 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1122 0.5662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3978 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3978 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1122 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8267 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6833 0.5662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9688 0.1537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9688 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6833 -1.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1122 -1.6559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6281 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3425 0.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0570 0.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0570 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3425 1.8538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6281 1.4413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7405 1.8359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6833 -1.8538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2783 0.5523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.7185 0.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8845 -0.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4718 -1.0226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6735 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1200 -1.0403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2926 -0.3255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0910 -0.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2480 0.0512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3677 -0.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9205 -0.7635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8918 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7949 0.4082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2069 1.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7949 1.8352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0307 1.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4427 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2666 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6791 1.1226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5041 1.1226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9166 0.4082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5041 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6791 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.7405 0.4082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9160 1.8361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
14 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
25 9 1 0 0 0 0
31 24 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
39 44 1 0 0 0 0
32 28 1 0 0 0 0
S SKP 8
ID FL3FACCS0058
KNApSAcK_ID C00014100
NAME Isoorientin 6''-O-caffeate
CAS_RN 172377-88-7
FORMULA C30H26O14
EXACTMASS 610.13225554
AVERAGEMASS 610.51904
SMILES c(c5)c(cc(O)c(O)5)C(=C4)Oc(c3)c(C(=O)4)c(c(c3O)C(C1O)OC(COC(=O)C=Cc(c2)ccc(O)c2O)C(C1O)O)O
M END