Mol:FL3FACCS0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6029 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6029 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6029 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6029 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0466 -0.0300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0466 -0.0300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5097 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5097 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5097 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5097 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0466 1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0466 1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0660 -0.0300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0660 -0.0300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6223 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6223 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6223 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6223 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0660 1.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0660 1.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0466 -0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0466 -0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1590 1.2546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1590 1.2546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1829 1.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1829 1.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7117 0.9722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7117 0.9722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2404 1.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2404 1.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2404 1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2404 1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7117 2.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7117 2.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1829 1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1829 1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7691 2.1933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7691 2.1933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8056 0.3147 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.8056 0.3147 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.4137 -0.3246 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4137 -0.3246 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.8981 -0.0153 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8981 -0.0153 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.2381 -0.1390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.2381 -0.1390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6919 0.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6919 0.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2487 0.0879 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2487 0.0879 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.3938 0.3662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3938 0.3662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1487 -0.6672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1487 -0.6672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5232 -0.6647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5232 -0.6647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7117 2.8039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7117 2.8039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5232 -1.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5232 -1.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1565 -1.8737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1565 -1.8737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1148 -1.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1148 -1.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6663 -1.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6663 -1.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2178 -1.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2178 -1.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2178 -2.4855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2178 -2.4855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6663 -2.8039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6663 -2.8039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1148 -2.4855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1148 -2.4855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7691 -2.8038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7691 -2.8038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0660 -0.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0660 -0.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4498 0.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4498 0.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4049 1.2712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4049 1.2712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 3 11 1 0 0 0 0 | + | 3 11 1 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 2 1 0 0 0 0 | + | 23 2 1 0 0 0 0 |
| − | 17 29 1 0 0 0 0 | + | 17 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 7 39 2 0 0 0 0 | + | 7 39 2 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -2.4498 0.975 | + | M SVB 1 44 -2.4498 0.975 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACCS0034 | + | ID FL3FACCS0034 |
| − | KNApSAcK_ID C00006257 | + | KNApSAcK_ID C00006257 |
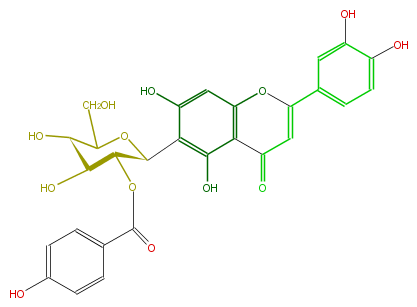
| − | NAME Isoorientin 2''-p-hydroxybenzoate | + | NAME Isoorientin 2''-p-hydroxybenzoate |
| − | CAS_RN 67308-38-7 | + | CAS_RN 67308-38-7 |
| − | FORMULA C28H24O13 | + | FORMULA C28H24O13 |
| − | EXACTMASS 568.121690854 | + | EXACTMASS 568.121690854 |
| − | AVERAGEMASS 568.48236 | + | AVERAGEMASS 568.48236 |
| − | SMILES c(c5O)(ccc(c5)C(O1)=CC(=O)c(c2O)c1cc(c2[C@@H]([C@H]3OC(c(c4)ccc(c4)O)=O)OC([C@H](O)[C@@H]3O)CO)O)O | + | SMILES c(c5O)(ccc(c5)C(O1)=CC(=O)c(c2O)c1cc(c2[C@@H]([C@H]3OC(c(c4)ccc(c4)O)=O)OC([C@H](O)[C@@H]3O)CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-0.6029 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6029 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0466 -0.0300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5097 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5097 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0466 1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0660 -0.0300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6223 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6223 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0660 1.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0466 -0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1590 1.2546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1829 1.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7117 0.9722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2404 1.2775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2404 1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7117 2.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1829 1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7691 2.1933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8056 0.3147 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.4137 -0.3246 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.8981 -0.0153 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.2381 -0.1390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6919 0.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2487 0.0879 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.3938 0.3662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1487 -0.6672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5232 -0.6647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7117 2.8039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5232 -1.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1565 -1.8737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1148 -1.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6663 -1.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2178 -1.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2178 -2.4855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6663 -2.8039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1148 -2.4855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7691 -2.8038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0660 -0.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4498 0.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4049 1.2712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
3 11 1 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 2 1 0 0 0 0
17 29 1 0 0 0 0
28 30 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 32 1 0 0 0 0
35 38 1 0 0 0 0
7 39 2 0 0 0 0
25 40 1 0 0 0 0
40 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -2.4498 0.975
S SKP 8
ID FL3FACCS0034
KNApSAcK_ID C00006257
NAME Isoorientin 2''-p-hydroxybenzoate
CAS_RN 67308-38-7
FORMULA C28H24O13
EXACTMASS 568.121690854
AVERAGEMASS 568.48236
SMILES c(c5O)(ccc(c5)C(O1)=CC(=O)c(c2O)c1cc(c2[C@@H]([C@H]3OC(c(c4)ccc(c4)O)=O)OC([C@H](O)[C@@H]3O)CO)O)O
M END