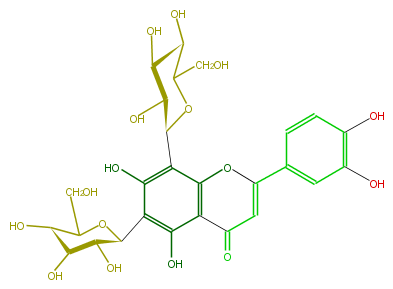
Mol:FL3FACCS0032
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1085 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1085 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1085 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1085 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5522 -1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5522 -1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0041 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0041 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0041 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0041 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5522 -0.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5522 -0.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5604 -1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5604 -1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1167 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1167 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1167 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1167 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5604 -0.5374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5604 -0.5374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5604 -2.3230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5604 -2.3230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6646 -0.5375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6646 -0.5375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7347 -0.5169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7347 -0.5169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3209 -0.8554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3209 -0.8554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9071 -0.5169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9071 -0.5169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9071 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9071 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3209 0.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3209 0.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7347 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7347 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4929 0.4982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4929 0.4982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3274 1.9408 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.3274 1.9408 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.9330 1.4820 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9330 1.4820 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6761 0.8214 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6761 0.8214 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6692 0.1839 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6692 0.1839 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2059 0.6471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2059 0.6471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5110 1.2251 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.5110 1.2251 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.4892 2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4892 2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9678 2.1972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9678 2.1972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3116 0.4429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3116 0.4429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5522 -2.4643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5522 -2.4643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9791 -1.7485 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.9791 -1.7485 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.6079 -2.2384 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6079 -2.2384 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.0734 -2.0306 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.0734 -2.0306 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5577 -2.0250 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5577 -2.0250 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.9325 -1.6501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9325 -1.6501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4001 -1.8969 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.4001 -1.8969 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.5700 -1.6967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5700 -1.6967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9583 -2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9583 -2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7672 -2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7672 -2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4929 -0.8552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4929 -0.8552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6013 -1.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6013 -1.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5564 -0.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5564 -0.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0044 1.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0044 1.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7189 1.1101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7189 1.1101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 2 1 0 0 0 0 | + | 33 2 1 0 0 0 0 |
| − | 15 39 1 0 0 0 0 | + | 15 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 0.0044 1.5226 | + | M SVB 2 46 0.0044 1.5226 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -2.6013 -1.0089 | + | M SVB 1 44 -2.6013 -1.0089 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACCS0032 | + | ID FL3FACCS0032 |
| − | KNApSAcK_ID C00006231 | + | KNApSAcK_ID C00006231 |
| − | NAME Lucenin 2 | + | NAME Lucenin 2 |
| − | CAS_RN 29428-58-8 | + | CAS_RN 29428-58-8 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES O[C@@H]([C@H]1O)[C@H](c(c4O)c(c(C(=O)2)c(c4[C@H](O5)[C@H]([C@H]([C@H](C5CO)O)O)O)OC(c(c3)ccc(O)c3O)=C2)O)OC(CO)[C@@H]1O | + | SMILES O[C@@H]([C@H]1O)[C@H](c(c4O)c(c(C(=O)2)c(c4[C@H](O5)[C@H]([C@H]([C@H](C5CO)O)O)O)OC(c(c3)ccc(O)c3O)=C2)O)OC(CO)[C@@H]1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.1085 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1085 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5522 -1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0041 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0041 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5522 -0.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5604 -1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1167 -1.5010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1167 -0.8586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5604 -0.5374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5604 -2.3230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6646 -0.5375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7347 -0.5169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3209 -0.8554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9071 -0.5169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9071 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3209 0.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7347 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4929 0.4982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3274 1.9408 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.9330 1.4820 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6761 0.8214 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6692 0.1839 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2059 0.6471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5110 1.2251 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.4892 2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9678 2.1972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3116 0.4429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5522 -2.4643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9791 -1.7485 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.6079 -2.2384 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.0734 -2.0306 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5577 -2.0250 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.9325 -1.6501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4001 -1.8969 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.5700 -1.6967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9583 -2.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7672 -2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4929 -0.8552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6013 -1.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5564 -0.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0044 1.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7189 1.1101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 2 1 0 0 0 0
15 39 1 0 0 0 0
35 40 1 0 0 0 0
40 41 1 0 0 0 0
25 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 0.0044 1.5226
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -2.6013 -1.0089
S SKP 8
ID FL3FACCS0032
KNApSAcK_ID C00006231
NAME Lucenin 2
CAS_RN 29428-58-8
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES O[C@@H]([C@H]1O)[C@H](c(c4O)c(c(C(=O)2)c(c4[C@H](O5)[C@H]([C@H]([C@H](C5CO)O)O)O)OC(c(c3)ccc(O)c3O)=C2)O)OC(CO)[C@@H]1O
M END