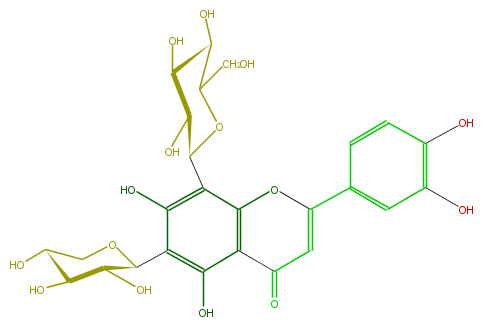
Mol:FL3FACCS0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4901 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4901 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4901 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4901 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7756 -2.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7756 -2.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0612 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0612 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0612 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0612 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7756 -0.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7756 -0.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6532 -2.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6532 -2.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3676 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3676 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3676 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3676 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6532 -0.5803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6532 -0.5803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6532 -2.8734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6532 -2.8734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2042 -0.5804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2042 -0.5804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6049 2.3310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6049 2.3310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3826 1.7418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3826 1.7418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0527 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0527 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0438 0.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0438 0.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4488 0.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4488 0.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8405 1.4119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8405 1.4119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7651 2.9292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7651 2.9292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3826 2.3990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3826 2.3990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4665 0.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4665 0.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7756 -3.0549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7756 -3.0549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1613 -0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1613 -0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9141 -0.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9141 -0.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6670 -0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6670 -0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6670 0.3153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6670 0.3153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9141 0.7499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9141 0.7499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1613 0.3153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1613 0.3153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4194 0.7497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4194 0.7497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9303 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9303 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4537 -2.4320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4537 -2.4320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7673 -2.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7673 -2.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1049 -2.1579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1049 -2.1579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5862 -1.6765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5862 -1.6765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1867 -1.9934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1867 -1.9934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4194 -2.0852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4194 -2.0852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0125 -2.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0125 -2.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0263 -2.5929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0263 -2.5929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4194 -0.9884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4194 -0.9884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3096 1.9428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3096 1.9428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3325 3.0549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3325 3.0549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 9 1 0 0 0 0 | + | 23 9 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 2 1 0 0 0 0 | + | 33 2 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 18 40 1 0 0 0 0 | + | 18 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 45 -0.5309 -0.5309 | + | M SBV 1 45 -0.5309 -0.5309 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACCS0018 | + | ID FL3FACCS0018 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES C(C5O)OC(C(C5O)O)c(c3O)c(c(c1c3C(O4)C(C(C(O)C4CO)O)O)C(C=C(c(c2)ccc(O)c2O)O1)=O)O | + | SMILES C(C5O)OC(C(C5O)O)c(c3O)c(c(c1c3C(O4)C(C(C(O)C4CO)O)O)C(C=C(c(c2)ccc(O)c2O)O1)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.4901 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4901 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7756 -2.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0612 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0612 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7756 -0.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6532 -2.2302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3676 -1.8178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3676 -0.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6532 -0.5803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6532 -2.8734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2042 -0.5804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6049 2.3310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3826 1.7418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0527 0.8933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0438 0.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4488 0.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8405 1.4119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7651 2.9292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3826 2.3990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4665 0.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7756 -3.0549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1613 -0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9141 -0.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6670 -0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6670 0.3153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9141 0.7499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1613 0.3153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4194 0.7497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9303 -1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4537 -2.4320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7673 -2.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1049 -2.1579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5862 -1.6765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1867 -1.9934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4194 -2.0852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0125 -2.5817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0263 -2.5929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4194 -0.9884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3096 1.9428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3325 3.0549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
23 9 1 0 0 0 0
26 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 2 1 0 0 0 0
25 39 1 0 0 0 0
40 41 1 0 0 0 0
18 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 CH2OH
M SBV 1 45 -0.5309 -0.5309
S SKP 5
ID FL3FACCS0018
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES C(C5O)OC(C(C5O)O)c(c3O)c(c(c1c3C(O4)C(C(C(O)C4CO)O)O)C(C=C(c(c2)ccc(O)c2O)O1)=O)O
M END