Mol:FL3FABGS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 38 0 0 0 0 0 0 0 0999 V2000 | + | 35 38 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2634 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2634 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2634 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2634 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1877 -1.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1877 -1.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6387 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6387 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6387 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6387 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1877 -0.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1877 -0.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0898 -1.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0898 -1.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5409 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5409 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5409 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5409 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0898 -0.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0898 -0.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0898 -1.5371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0898 -1.5371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9918 -0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9918 -0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4515 -0.3548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4515 -0.3548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9112 -0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9112 -0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9112 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9112 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4515 0.7068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4515 0.7068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9918 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9918 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1877 -1.5374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1877 -1.5374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0309 0.0392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0309 0.0392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4443 -0.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4443 -0.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9714 -0.8095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9714 -0.8095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2906 -0.5447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2906 -0.5447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6336 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6336 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1110 -0.0601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1110 -0.0601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7067 -0.3745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7067 -0.3745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0988 -0.5633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0988 -0.5633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7085 -0.8454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7085 -0.8454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9004 -1.1996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9004 -1.1996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2157 0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2157 0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2157 0.6304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2157 0.6304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7775 0.9547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7775 0.9547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3236 0.6394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3236 0.6394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7775 1.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7775 1.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6092 0.7393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6092 0.7393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3236 0.3268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3236 0.3268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 3 1 0 0 0 0 | + | 18 3 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 15 34 1 0 0 0 0 | + | 15 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 34 35 | + | M SAL 1 2 34 35 |
| − | M SBL 1 1 37 | + | M SBL 1 1 37 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 37 -7.0646 4.8464 | + | M SBV 1 37 -7.0646 4.8464 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FABGS0013 | + | ID FL3FABGS0013 |
| − | KNApSAcK_ID C00004245 | + | KNApSAcK_ID C00004245 |
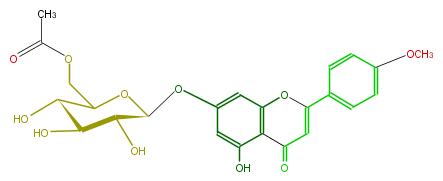
| − | NAME Acacetin 7-(6''-acetylglucoside) | + | NAME Acacetin 7-(6''-acetylglucoside) |
| − | CAS_RN 76410-61-2 | + | CAS_RN 76410-61-2 |
| − | FORMULA C24H24O11 | + | FORMULA C24H24O11 |
| − | EXACTMASS 488.13186161 | + | EXACTMASS 488.13186161 |
| − | AVERAGEMASS 488.44076 | + | AVERAGEMASS 488.44076 |
| − | SMILES Oc(c1)c(C(=O)4)c(OC(=C4)c(c3)ccc(c3)OC)cc1OC(C2O)OC(COC(C)=O)C(C2O)O | + | SMILES Oc(c1)c(C(=O)4)c(OC(=C4)c(c3)ccc(c3)OC)cc1OC(C2O)OC(COC(C)=O)C(C2O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 38 0 0 0 0 0 0 0 0999 V2000
-0.2634 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2634 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1877 -1.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6387 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6387 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1877 -0.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0898 -1.1310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5409 -0.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5409 -0.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0898 -0.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0898 -1.5371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9918 -0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4515 -0.3548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9112 -0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9112 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4515 0.7068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9918 0.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1877 -1.5374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0309 0.0392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4443 -0.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9714 -0.8095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2906 -0.5447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6336 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1110 -0.0601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7067 -0.3745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0988 -0.5633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7085 -0.8454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9004 -1.1996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2157 0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2157 0.6304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7775 0.9547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3236 0.6394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7775 1.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6092 0.7393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3236 0.3268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 3 1 0 0 0 0
19 1 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 19 1 0 0 0 0
25 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
15 34 1 0 0 0 0
34 35 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 34 35
M SBL 1 1 37
M SMT 1 OCH3
M SBV 1 37 -7.0646 4.8464
S SKP 8
ID FL3FABGS0013
KNApSAcK_ID C00004245
NAME Acacetin 7-(6''-acetylglucoside)
CAS_RN 76410-61-2
FORMULA C24H24O11
EXACTMASS 488.13186161
AVERAGEMASS 488.44076
SMILES Oc(c1)c(C(=O)4)c(OC(=C4)c(c3)ccc(c3)OC)cc1OC(C2O)OC(COC(C)=O)C(C2O)O
M END