Mol:FL3FABGS0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3604 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3604 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3604 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3604 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3406 -1.0764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3406 -1.0764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0417 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0417 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0417 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0417 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3406 0.5425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3406 0.5425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7426 -1.0764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7426 -1.0764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4437 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4437 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4437 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4437 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7426 0.5425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7426 0.5425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7426 -1.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7426 -1.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1444 0.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1444 0.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8589 0.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8589 0.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5734 0.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5734 0.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5734 1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5734 1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8589 1.7799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8589 1.7799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1444 1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1444 1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0612 0.5424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0612 0.5424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3406 -1.9118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3406 -1.9118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3349 0.7217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3349 0.7217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6938 -0.1245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6938 -0.1245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7707 0.2344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7707 0.2344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8798 0.2440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8798 0.2440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5271 0.8915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5271 0.8915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4701 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4701 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1176 0.3961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1176 0.3961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5794 -0.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5794 -0.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8806 -0.4304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8806 -0.4304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8566 -1.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8566 -1.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1995 -1.1786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1995 -1.1786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3611 -1.8302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3611 -1.8302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5624 -1.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5624 -1.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7043 -1.0046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7043 -1.0046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4972 -0.5467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4972 -0.5467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3205 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3205 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1186 -2.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1186 -2.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5055 -1.5847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5055 -1.5847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2548 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2548 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2766 -0.5794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2766 -0.5794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3981 -1.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3981 -1.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2877 1.7799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2877 1.7799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3981 1.1387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3981 1.1387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9260 1.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9260 1.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9517 1.1090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9517 1.1090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4114 2.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4114 2.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 15 41 1 0 0 0 0 | + | 15 41 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 25 43 1 0 0 0 0 | + | 25 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 38 39 40 | + | M SAL 1 3 38 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 44 0.9343 -0.4491 | + | M SBV 1 44 0.9343 -0.4491 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 46 -0.7143 -0.4125 | + | M SBV 2 46 -0.7143 -0.4125 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 3 43 44 45 | + | M SAL 3 3 43 44 45 |
| − | M SBL 3 1 49 | + | M SBL 3 1 49 |
| − | M SMT 3 ^ COOH | + | M SMT 3 ^ COOH |
| − | M SBV 3 49 0.4558 -0.5562 | + | M SBV 3 49 0.4558 -0.5562 |
| − | S SKP 5 | + | S SKP 5 |
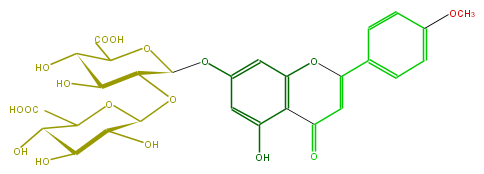
| − | ID FL3FABGS0007 | + | ID FL3FABGS0007 |
| − | FORMULA C28H28O17 | + | FORMULA C28H28O17 |
| − | EXACTMASS 636.1326494699999 | + | EXACTMASS 636.1326494699999 |
| − | AVERAGEMASS 636.51172 | + | AVERAGEMASS 636.51172 |
| − | SMILES O(C)c(c5)ccc(c5)C(=C4)Oc(c(C4=O)3)cc(cc(O)3)OC(C1OC(O2)C(O)C(O)C(C(C(O)=O)2)O)OC(C(O)=O)C(C1O)O | + | SMILES O(C)c(c5)ccc(c5)C(=C4)Oc(c(C4=O)3)cc(cc(O)3)OC(C1OC(O2)C(O)C(O)C(C(C(O)=O)2)O)OC(C(O)=O)C(C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-0.3604 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3604 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3406 -1.0764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0417 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0417 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3406 0.5425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7426 -1.0764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4437 -0.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4437 0.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7426 0.5425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7426 -1.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1444 0.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8589 0.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5734 0.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5734 1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8589 1.7799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1444 1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0612 0.5424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3406 -1.9118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3349 0.7217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6938 -0.1245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7707 0.2344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8798 0.2440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5271 0.8915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4701 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1176 0.3961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5794 -0.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8806 -0.4304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8566 -1.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1995 -1.1786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3611 -1.8302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5624 -1.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7043 -1.0046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4972 -0.5467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3205 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1186 -2.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5055 -1.5847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2548 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2766 -0.5794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3981 -1.1573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2877 1.7799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3981 1.1387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9260 1.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9517 1.1090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4114 2.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
32 37 1 0 0 0 0
29 30 1 0 0 0 0
33 28 1 0 0 0 0
23 18 1 0 0 0 0
38 39 2 0 0 0 0
38 40 1 0 0 0 0
35 38 1 0 0 0 0
41 42 1 0 0 0 0
15 41 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
25 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 38 39 40
M SBL 1 1 44
M SMT 1 ^ COOH
M SBV 1 44 0.9343 -0.4491
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 OCH3
M SBV 2 46 -0.7143 -0.4125
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 3 43 44 45
M SBL 3 1 49
M SMT 3 ^ COOH
M SBV 3 49 0.4558 -0.5562
S SKP 5
ID FL3FABGS0007
FORMULA C28H28O17
EXACTMASS 636.1326494699999
AVERAGEMASS 636.51172
SMILES O(C)c(c5)ccc(c5)C(=C4)Oc(c(C4=O)3)cc(cc(O)3)OC(C1OC(O2)C(O)C(O)C(C(C(O)=O)2)O)OC(C(O)=O)C(C1O)O
M END