Mol:FL3FABCS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2284 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2284 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2284 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2284 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6721 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6721 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1158 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1158 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1158 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1158 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6721 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6721 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4405 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4405 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9968 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9968 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9968 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9968 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4405 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4405 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4405 -1.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4405 -1.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6721 -1.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6721 -1.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6766 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6766 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2628 0.1356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2628 0.1356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8491 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8491 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8491 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8491 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2628 1.4894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2628 1.4894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6766 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6766 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7818 -0.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7818 -0.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2662 -1.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2662 -1.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7505 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7505 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0493 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0493 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5443 -0.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5443 -0.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1012 -1.0020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1012 -1.0020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2936 -1.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2936 -1.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9085 -1.4970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9085 -1.4970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8007 0.3803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8007 0.3803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4412 -1.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4412 -1.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1372 -0.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1372 -0.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8364 -0.7710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8364 -0.7710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4148 -0.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4148 -0.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9968 -0.7710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9968 -0.7710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2977 -0.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2977 -0.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7192 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7192 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5785 -0.3997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5785 -0.3997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9574 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9574 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7318 -0.5006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7318 -0.5006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5791 1.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5791 1.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2936 1.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2936 1.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3498 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3498 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0643 -1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0643 -1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4542 -0.6157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4542 -0.6157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0375 -0.0324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0375 -0.0324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 2 22 1 0 0 0 0 | + | 2 22 1 0 0 0 0 |
| − | 1 27 1 0 0 0 0 | + | 1 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 30 8 1 0 0 0 0 | + | 30 8 1 0 0 0 0 |
| − | 16 38 1 0 0 0 0 | + | 16 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 24 42 1 0 0 0 0 | + | 24 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 42 | + | M SBL 1 1 42 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 42 -4.2067 9.4911 | + | M SBV 1 42 -4.2067 9.4911 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 44 | + | M SBL 2 1 44 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 44 -4.5838 9.0278 | + | M SBV 2 44 -4.5838 9.0278 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 42 43 | + | M SAL 3 2 42 43 |
| − | M SBL 3 1 46 | + | M SBL 3 1 46 |
| − | M SMT 3 ^CH2OH | + | M SMT 3 ^CH2OH |
| − | M SBV 3 46 -5.2898 9.3874 | + | M SBV 3 46 -5.2898 9.3874 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FABCS0006 | + | ID FL3FABCS0006 |
| − | KNApSAcK_ID C00006286 | + | KNApSAcK_ID C00006286 |
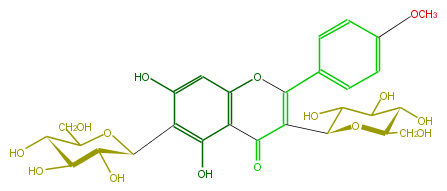
| − | NAME 3,6-Di-C-glucopyranosylacacetin | + | NAME 3,6-Di-C-glucopyranosylacacetin |
| − | CAS_RN 98891-91-9 | + | CAS_RN 98891-91-9 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES C(c(c(O)2)c(c(C3=O)c(OC(c(c5)ccc(OC)c5)=C(C(C(O)4)OC(C(O)C4O)CO)3)c2)O)(O1)C(O)C(C(C1CO)O)O | + | SMILES C(c(c(O)2)c(c(C3=O)c(OC(c(c5)ccc(OC)c5)=C(C(C(O)4)OC(C(O)C4O)CO)3)c2)O)(O1)C(O)C(C(C1CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.2284 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2284 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6721 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1158 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1158 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6721 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4405 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9968 -0.5925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9968 0.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4405 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4405 -1.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6721 -1.5558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6766 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2628 0.1356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8491 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8491 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2628 1.4894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6766 1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7818 -0.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2662 -1.4145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7505 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0493 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5443 -0.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1012 -1.0020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2936 -1.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9085 -1.4970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8007 0.3803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4412 -1.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1372 -0.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8364 -0.7710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4148 -0.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9968 -0.7710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2977 -0.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7192 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5785 -0.3997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9574 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7318 -0.5006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5791 1.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2936 1.2285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3498 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0643 -1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4542 -0.6157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0375 -0.0324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
2 22 1 0 0 0 0
1 27 1 0 0 0 0
21 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
30 8 1 0 0 0 0
16 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
40 41 1 0 0 0 0
24 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 42
M SMT 1 OCH3
M SBV 1 42 -4.2067 9.4911
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 44
M SMT 2 CH2OH
M SBV 2 44 -4.5838 9.0278
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 42 43
M SBL 3 1 46
M SMT 3 ^CH2OH
M SBV 3 46 -5.2898 9.3874
S SKP 8
ID FL3FABCS0006
KNApSAcK_ID C00006286
NAME 3,6-Di-C-glucopyranosylacacetin
CAS_RN 98891-91-9
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES C(c(c(O)2)c(c(C3=O)c(OC(c(c5)ccc(OC)c5)=C(C(C(O)4)OC(C(O)C4O)CO)3)c2)O)(O1)C(O)C(C(C1CO)O)O
M END