Mol:FL3FAAGS0025
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2428 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2428 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2428 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2428 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4582 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4582 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1592 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1592 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1592 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1592 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4582 0.9350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4582 0.9350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8602 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8602 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5612 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5612 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5612 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5612 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8602 0.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8602 0.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8602 -1.5344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8602 -1.5344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2620 0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2620 0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9765 0.5224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9765 0.5224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6908 0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6908 0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6908 1.7599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6908 1.7599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9765 2.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9765 2.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2620 1.7599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2620 1.7599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9436 0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9436 0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4582 -1.4930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4582 -1.4930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4052 2.1722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4052 2.1722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9625 0.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9625 0.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3777 0.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3777 0.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5356 0.4429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5356 0.4429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7231 0.4516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7231 0.4516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3135 1.0421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3135 1.0421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1738 0.7333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1738 0.7333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6067 0.7146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6067 0.7146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1581 0.2368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1581 0.2368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7363 -0.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7363 -0.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7101 -2.1384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7101 -2.1384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2909 -1.4816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2909 -1.4816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3985 -1.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3985 -1.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8329 -1.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8329 -1.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1337 -0.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1337 -0.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9403 -0.5230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9403 -0.5230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5309 -1.2205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5309 -1.2205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9905 -2.1723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9905 -2.1723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8851 -1.1815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8851 -1.1815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4837 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4837 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4052 -1.2369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4052 -1.2369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3625 -1.7913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3625 -1.7913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6987 1.2583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6987 1.2583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6344 1.2583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6344 1.2583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2292 2.0716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2292 2.0716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 26 42 1 0 0 0 0 | + | 26 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 39 40 41 | + | M SAL 1 3 39 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 45 0.9528 -0.1461 | + | M SBV 1 45 0.9528 -0.1461 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 42 43 44 | + | M SAL 2 3 42 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 ^ COOH | + | M SMT 2 ^ COOH |
| − | M SBV 2 48 0.5250 -0.5250 | + | M SBV 2 48 0.5250 -0.5250 |
| − | S SKP 5 | + | S SKP 5 |
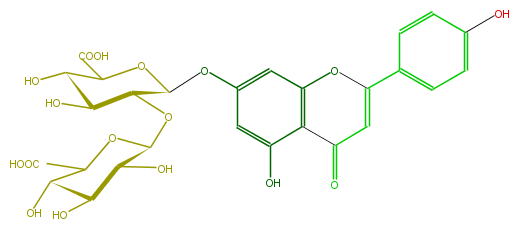
| − | ID FL3FAAGS0025 | + | ID FL3FAAGS0025 |
| − | FORMULA C27H26O17 | + | FORMULA C27H26O17 |
| − | EXACTMASS 622.116999406 | + | EXACTMASS 622.116999406 |
| − | AVERAGEMASS 622.48514 | + | AVERAGEMASS 622.48514 |
| − | SMILES c(c5O)c(cc(c45)OC(=CC4=O)c(c3)ccc(c3)O)OC(O1)C(OC(O2)C(O)C(C(C2C(O)=O)O)O)C(O)C(C1C(O)=O)O | + | SMILES c(c5O)c(cc(c45)OC(=CC4=O)c(c3)ccc(c3)O)OC(O1)C(OC(O2)C(O)C(C(C2C(O)=O)O)O)C(O)C(C1C(O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.2428 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2428 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4582 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1592 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1592 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4582 0.9350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8602 -0.6839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5612 -0.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5612 0.5303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8602 0.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8602 -1.5344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2620 0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9765 0.5224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6908 0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6908 1.7599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9765 2.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2620 1.7599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9436 0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4582 -1.4930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4052 2.1722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9625 0.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3777 0.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5356 0.4429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7231 0.4516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3135 1.0421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1738 0.7333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6067 0.7146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1581 0.2368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7363 -0.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7101 -2.1384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2909 -1.4816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3985 -1.8577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8329 -1.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1337 -0.7392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9403 -0.5230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5309 -1.2205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9905 -2.1723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8851 -1.1815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4837 -1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4052 -1.2369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3625 -1.7913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6987 1.2583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6344 1.2583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2292 2.0716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
30 31 1 0 0 0 0
34 29 1 0 0 0 0
24 18 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
36 39 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
26 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 39 40 41
M SBL 1 1 45
M SMT 1 ^ COOH
M SBV 1 45 0.9528 -0.1461
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 42 43 44
M SBL 2 1 48
M SMT 2 ^ COOH
M SBV 2 48 0.5250 -0.5250
S SKP 5
ID FL3FAAGS0025
FORMULA C27H26O17
EXACTMASS 622.116999406
AVERAGEMASS 622.48514
SMILES c(c5O)c(cc(c45)OC(=CC4=O)c(c3)ccc(c3)O)OC(O1)C(OC(O2)C(O)C(C(C2C(O)=O)O)O)C(O)C(C1C(O)=O)O
M END