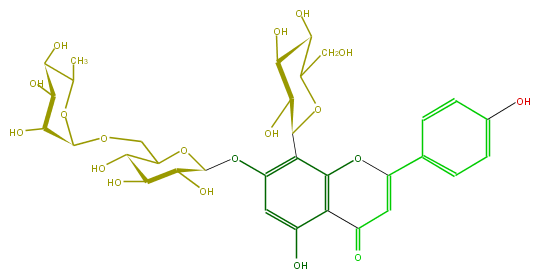
Mol:FL3FAADS0028
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 52 57 0 0 0 0 0 0 0 0999 V2000 | + | 52 57 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0958 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0958 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0958 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0958 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6186 -2.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6186 -2.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3331 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3331 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3331 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3331 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6186 -0.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6186 -0.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0475 -2.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0475 -2.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7620 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7620 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7620 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7620 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0475 -0.4548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0475 -0.4548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0475 -2.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0475 -2.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8100 -0.4549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8100 -0.4549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9544 2.3536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9544 2.3536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1766 1.7643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1766 1.7643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5066 0.9159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5066 0.9159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5154 0.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5154 0.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1105 0.6921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1105 0.6921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7187 1.4344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7187 1.4344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6707 2.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6707 2.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1539 2.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1539 2.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0400 0.1396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0400 0.1396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6186 -2.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6186 -2.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5557 -0.4284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5557 -0.4284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3086 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3086 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0614 -0.4284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0614 -0.4284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0614 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0614 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3086 0.8756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3086 0.8756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5557 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5557 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8139 0.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8139 0.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3340 -0.3936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3340 -0.3936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8572 -1.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8572 -1.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1708 -0.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1708 -0.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5084 -0.7489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5084 -0.7489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9898 -0.2674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9898 -0.2674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5903 -0.5844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5903 -0.5844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9100 -0.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9100 -0.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5372 -0.9987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5372 -0.9987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5492 -1.2115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5492 -1.2115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9930 -0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9930 -0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2454 0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2454 0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5709 -0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5709 -0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7849 0.5795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7849 0.5795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5709 1.3330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5709 1.3330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2454 1.7225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2454 1.7225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0315 0.9735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0315 0.9735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9582 2.1720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9582 2.1720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5709 1.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5709 1.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5130 1.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5130 1.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8139 0.1486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8139 0.1486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8508 0.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8508 0.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2641 2.0153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2641 2.0153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1818 1.4855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1818 1.4855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 9 1 0 0 0 0 | + | 23 9 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 33 12 1 0 0 0 0 | + | 33 12 1 0 0 0 0 |
| − | 41 40 1 1 0 0 0 | + | 41 40 1 1 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 40 1 1 0 0 0 | + | 45 40 1 1 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 43 47 1 0 0 0 0 | + | 43 47 1 0 0 0 0 |
| − | 45 48 1 0 0 0 0 | + | 45 48 1 0 0 0 0 |
| − | 40 49 1 0 0 0 0 | + | 40 49 1 0 0 0 0 |
| − | 41 50 1 0 0 0 0 | + | 41 50 1 0 0 0 0 |
| − | 50 39 1 0 0 0 0 | + | 50 39 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 18 51 1 0 0 0 0 | + | 18 51 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 57 -0.5454 -0.5809 | + | M SBV 1 57 -0.5454 -0.5809 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAADS0028 | + | ID FL3FAADS0028 |
| − | FORMULA C33H40O19 | + | FORMULA C33H40O19 |
| − | EXACTMASS 740.216379098 | + | EXACTMASS 740.216379098 |
| − | AVERAGEMASS 740.6593 | + | AVERAGEMASS 740.6593 |
| − | SMILES OC(C(O)1)C(COC(C6O)OC(C(O)C(O)6)C)OC(Oc(c(C(C(O)5)OC(CO)C(O)C5O)4)cc(O)c(c42)C(=O)C=C(c(c3)ccc(c3)O)O2)C1O | + | SMILES OC(C(O)1)C(COC(C6O)OC(C(O)C(O)6)C)OC(Oc(c(C(C(O)5)OC(CO)C(O)C5O)4)cc(O)c(c42)C(=O)C=C(c(c3)ccc(c3)O)O2)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
52 57 0 0 0 0 0 0 0 0999 V2000
-0.0958 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0958 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6186 -2.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3331 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3331 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6186 -0.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0475 -2.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7620 -1.6922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7620 -0.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0475 -0.4548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0475 -2.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8100 -0.4549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9544 2.3536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1766 1.7643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5066 0.9159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5154 0.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1105 0.6921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7187 1.4344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6707 2.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1539 2.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0400 0.1396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6186 -2.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5557 -0.4284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3086 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0614 -0.4284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0614 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3086 0.8756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5557 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8139 0.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3340 -0.3936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8572 -1.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1708 -0.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5084 -0.7489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9898 -0.2674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5903 -0.5844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9100 -0.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5372 -0.9987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5492 -1.2115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9930 -0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2454 0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5709 -0.1694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7849 0.5795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5709 1.3330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2454 1.7225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0315 0.9735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9582 2.1720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5709 1.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5130 1.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8139 0.1486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8508 0.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2641 2.0153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1818 1.4855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
23 9 1 0 0 0 0
26 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
35 39 1 0 0 0 0
33 12 1 0 0 0 0
41 40 1 1 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 1 0 0 0
45 40 1 1 0 0 0
44 46 1 0 0 0 0
43 47 1 0 0 0 0
45 48 1 0 0 0 0
40 49 1 0 0 0 0
41 50 1 0 0 0 0
50 39 1 0 0 0 0
51 52 1 0 0 0 0
18 51 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 CH2OH
M SBV 1 57 -0.5454 -0.5809
S SKP 5
ID FL3FAADS0028
FORMULA C33H40O19
EXACTMASS 740.216379098
AVERAGEMASS 740.6593
SMILES OC(C(O)1)C(COC(C6O)OC(C(O)C(O)6)C)OC(Oc(c(C(C(O)5)OC(CO)C(O)C5O)4)cc(O)c(c42)C(=O)C=C(c(c3)ccc(c3)O)O2)C1O
M END