Mol:FL3FAADS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0626 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0626 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0626 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0626 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3480 -0.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3480 -0.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3665 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3665 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3665 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3665 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3480 1.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3480 1.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0810 -0.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0810 -0.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7956 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7956 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7956 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7956 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0810 1.1437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0810 1.1437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0810 -1.1498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0810 -1.1498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3480 -1.3312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3480 -1.3312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6687 1.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6687 1.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4217 0.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4217 0.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1746 1.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1746 1.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1746 2.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1746 2.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4217 2.5802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4217 2.5802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6687 2.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6687 2.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9272 2.5799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9272 2.5799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7769 1.1435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7769 1.1435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3305 1.1514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3305 1.1514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8537 0.5220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8537 0.5220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1672 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1672 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5046 0.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5046 0.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9861 1.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9861 1.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5867 0.9607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5867 0.9607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9272 0.9546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9272 0.9546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5749 0.5669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5749 0.5669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6280 0.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6280 0.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3993 -2.2112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3993 -2.2112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0070 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0070 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5723 -1.0312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5723 -1.0312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7745 -0.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7745 -0.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1277 -0.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1277 -0.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5357 -1.5972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5357 -1.5972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9066 -2.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9066 -2.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6487 -2.1725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6487 -2.1725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8620 -2.1855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8620 -2.1855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6745 -1.3886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6745 -1.3886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1590 1.5747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1590 1.5747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1354 1.3005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1354 1.3005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 33 2 1 0 0 0 0 | + | 33 2 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 0.5724 -0.6140 | + | M SBV 1 45 0.5724 -0.6140 |
| − | S SKP 5 | + | S SKP 5 |
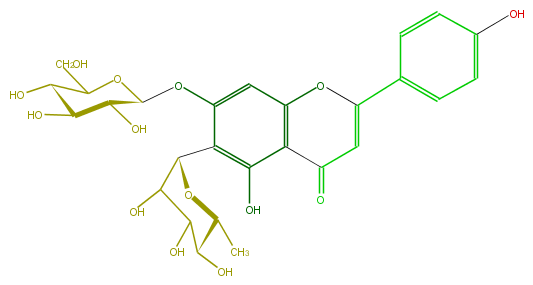
| − | ID FL3FAADS0008 | + | ID FL3FAADS0008 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES c(c1)(ccc(C(=C2)Oc(c4)c(c(O)c(c(OC(C5O)OC(C(O)C5O)CO)4)C(C(O)3)OC(C(O)C(O)3)C)C2=O)c1)O | + | SMILES c(c1)(ccc(C(=C2)Oc(c4)c(c(O)c(c(OC(C5O)OC(C(O)C5O)CO)4)C(C(O)3)OC(C(O)C(O)3)C)C2=O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.0626 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0626 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3480 -0.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3665 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3665 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3480 1.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0810 -0.5065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7956 -0.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7956 0.7311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0810 1.1437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0810 -1.1498 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3480 -1.3312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6687 1.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4217 0.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1746 1.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1746 2.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4217 2.5802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6687 2.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9272 2.5799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7769 1.1435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3305 1.1514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8537 0.5220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1672 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5046 0.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9861 1.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5867 0.9607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9272 0.9546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5749 0.5669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6280 0.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3993 -2.2112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0070 -1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5723 -1.0312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7745 -0.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1277 -0.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5357 -1.5972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9066 -2.5802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6487 -2.1725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8620 -2.1855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6745 -1.3886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1590 1.5747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1354 1.3005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 20 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
35 38 1 0 0 0 0
33 2 1 0 0 0 0
34 39 1 0 0 0 0
40 41 1 0 0 0 0
26 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 0.5724 -0.6140
S SKP 5
ID FL3FAADS0008
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES c(c1)(ccc(C(=C2)Oc(c4)c(c(O)c(c(OC(C5O)OC(C(O)C5O)CO)4)C(C(O)3)OC(C(O)C(O)3)C)C2=O)c1)O
M END