Mol:FL3FAADS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2219 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2219 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2219 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2219 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5074 -1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5074 -1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2070 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2070 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2070 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2070 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5074 0.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5074 0.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9215 -1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9215 -1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6360 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6360 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6360 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6360 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9215 0.1680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9215 0.1680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9215 -2.1251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9215 -2.1251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5074 -2.3066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5074 -2.3066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5092 0.3003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5092 0.3003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2620 -0.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2620 -0.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0149 0.3003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0149 0.3003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0149 1.1696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0149 1.1696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2620 1.6044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2620 1.6044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5092 1.1696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5092 1.1696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7239 1.6289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7239 1.6289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1300 -0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1300 -0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4677 -1.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4677 -1.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8055 -1.2946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8055 -1.2946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9049 -1.4006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9049 -1.4006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5407 -0.8179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5407 -0.8179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2558 -1.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2558 -1.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7239 -1.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7239 -1.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2927 -1.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2927 -1.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9569 0.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9569 0.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1592 -1.8193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1592 -1.8193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4490 0.3340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4490 0.3340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7744 -0.0554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7744 -0.0554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9885 0.6934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9885 0.6934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7744 1.4469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7744 1.4469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4490 1.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4490 1.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2350 1.0875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2350 1.0875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1411 2.3066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1411 2.3066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7744 1.9329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7744 1.9329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8816 1.4962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8816 1.4962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0174 0.3036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0174 0.3036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7091 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7091 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4584 0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4584 0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 31 28 1 0 0 0 0 | + | 31 28 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 0.4534 -0.4960 | + | M SBV 1 45 0.4534 -0.4960 |
| − | S SKP 5 | + | S SKP 5 |
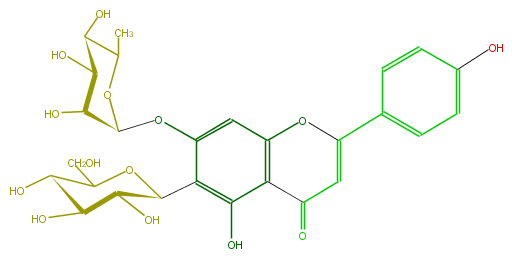
| − | ID FL3FAADS0006 | + | ID FL3FAADS0006 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES c(c1)(ccc(C(=C5)Oc(c(C5=O)4)cc(c(c4O)C(C(O)3)OC(C(O)C(O)3)CO)OC(O2)C(C(C(C2C)O)O)O)c1)O | + | SMILES c(c1)(ccc(C(=C5)Oc(c(C5=O)4)cc(c(c4O)C(C(O)3)OC(C(O)C(O)3)CO)OC(O2)C(C(C(C2C)O)O)O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.2219 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2219 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5074 -1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2070 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2070 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5074 0.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9215 -1.4819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6360 -1.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6360 -0.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9215 0.1680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9215 -2.1251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5074 -2.3066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5092 0.3003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2620 -0.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0149 0.3003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0149 1.1696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2620 1.6044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5092 1.1696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7239 1.6289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1300 -0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4677 -1.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8055 -1.2946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9049 -1.4006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5407 -0.8179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2558 -1.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7239 -1.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2927 -1.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9569 0.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1592 -1.8193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4490 0.3340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7744 -0.0554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9885 0.6934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7744 1.4469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4490 1.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2350 1.0875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1411 2.3066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7744 1.9329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8816 1.4962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0174 0.3036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7091 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4584 0.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
2 23 1 0 0 0 0
1 28 1 0 0 0 0
22 29 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
35 38 1 0 0 0 0
30 39 1 0 0 0 0
31 28 1 0 0 0 0
40 41 1 0 0 0 0
25 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 0.4534 -0.4960
S SKP 5
ID FL3FAADS0006
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES c(c1)(ccc(C(=C5)Oc(c(C5=O)4)cc(c(c4O)C(C(O)3)OC(C(O)C(O)3)CO)OC(O2)C(C(C(C2C)O)O)O)c1)O
M END