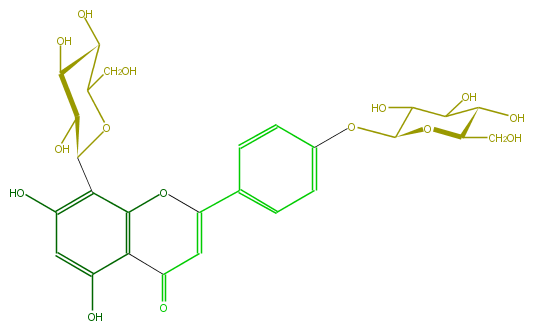
Mol:FL3FAADS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.2122 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2122 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2122 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2122 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4977 -2.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4977 -2.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7833 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7833 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7833 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7833 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4977 -0.5537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4977 -0.5537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0688 -2.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0688 -2.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3544 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3544 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3544 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3544 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0688 -0.5537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0688 -0.5537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0688 -2.8470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0688 -2.8470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9264 -0.5538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9264 -0.5538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5607 -0.5274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5607 -0.5274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1922 -0.9621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1922 -0.9621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9451 -0.5274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9451 -0.5274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9451 0.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9451 0.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1922 0.7766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1922 0.7766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5607 0.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5607 0.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6974 0.7763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6974 0.7763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3534 2.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3534 2.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1312 1.8129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1312 1.8129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8012 0.9645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8012 0.9645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7924 0.1458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7924 0.1458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1974 0.7407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1974 0.7407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5892 1.4830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5892 1.4830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7150 3.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7150 3.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1312 2.4793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1312 2.4793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1681 0.3290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1681 0.3290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4977 -3.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4977 -3.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9023 1.1404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9023 1.1404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5578 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5578 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2202 0.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2202 0.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8868 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8868 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2313 1.1404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2313 1.1404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5688 0.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5688 0.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3204 1.1404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3204 1.1404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9793 1.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9793 1.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9264 0.9542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9264 0.9542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5094 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5094 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4821 -0.7403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4821 -0.7403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1890 1.8833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1890 1.8833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2713 1.3535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2713 1.3535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 31 19 1 0 0 0 0 | + | 31 19 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -0.6227 0.0000 | + | M SBV 1 44 -0.6227 0.0000 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 -0.4003 -0.4003 | + | M SBV 2 46 -0.4003 -0.4003 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAADS0004 | + | ID FL3FAADS0004 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c5)(ccc(c5)OC(O4)C(O)C(O)C(O)C4CO)C(O3)=CC(=O)c(c13)c(O)cc(c(C(C(O)2)OC(C(O)C(O)2)CO)1)O | + | SMILES c(c5)(ccc(c5)OC(O4)C(O)C(O)C(O)C4CO)C(O3)=CC(=O)c(c13)c(O)cc(c(C(C(O)2)OC(C(O)C(O)2)CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-4.2122 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2122 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4977 -2.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7833 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7833 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4977 -0.5537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0688 -2.2037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3544 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3544 -0.9662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0688 -0.5537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0688 -2.8470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9264 -0.5538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5607 -0.5274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1922 -0.9621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9451 -0.5274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9451 0.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1922 0.7766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5607 0.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6974 0.7763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3534 2.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1312 1.8129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8012 0.9645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7924 0.1458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1974 0.7407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5892 1.4830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7150 3.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1312 2.4793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1681 0.3290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4977 -3.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9023 1.1404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5578 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2202 0.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8868 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2313 1.1404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5688 0.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3204 1.1404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9793 1.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9264 0.9542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5094 0.5438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4821 -0.7403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1890 1.8833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2713 1.3535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
31 19 1 0 0 0 0
39 40 1 0 0 0 0
33 39 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -0.6227 0.0000
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 -0.4003 -0.4003
S SKP 5
ID FL3FAADS0004
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c5)(ccc(c5)OC(O4)C(O)C(O)C(O)C4CO)C(O3)=CC(=O)c(c13)c(O)cc(c(C(C(O)2)OC(C(O)C(O)2)CO)1)O
M END