Mol:FL3FAACS0072
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.8813 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8813 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1669 0.5566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1669 0.5566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4524 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4524 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4524 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4524 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1669 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1669 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8813 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8813 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7379 0.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7379 0.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0234 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0234 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0234 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0234 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7379 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7379 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6476 0.5315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6476 0.5315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7379 -1.8329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7379 -1.8329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1669 -1.6655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1669 -1.6655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6830 -0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6830 -0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2704 -1.0982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2704 -1.0982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4721 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4721 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6786 -1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6786 -1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0911 -0.4011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0911 -0.4011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8895 -0.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8895 -0.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0525 -0.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0525 -0.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3981 -0.0297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3981 -0.0297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1662 -0.8667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1662 -0.8667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7191 -0.8391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7191 -0.8391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6435 -1.5290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6435 -1.5290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6631 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6631 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3775 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3775 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0920 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0920 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0920 1.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0920 1.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3775 1.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3775 1.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6631 1.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6631 1.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7072 1.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7072 1.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2240 0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2240 0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8114 -0.4217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8114 -0.4217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0131 -0.2125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0131 -0.2125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2196 -0.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2196 -0.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6321 0.2754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6321 0.2754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4305 0.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4305 0.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5935 0.4472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5935 0.4472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9391 0.6468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9391 0.6468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7072 -0.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7072 -0.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2601 -0.1626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2601 -0.1626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1845 -0.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1845 -0.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 5 13 2 0 0 0 0 | + | 5 13 2 0 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 15 16 1 1 0 0 0 | + | 15 16 1 1 0 0 0 |
| − | 17 16 1 1 0 0 0 | + | 17 16 1 1 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 15 23 1 0 0 0 0 | + | 15 23 1 0 0 0 0 |
| − | 16 24 1 0 0 0 0 | + | 16 24 1 0 0 0 0 |
| − | 9 17 1 0 0 0 0 | + | 9 17 1 0 0 0 0 |
| − | 1 25 1 0 0 0 0 | + | 1 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 21 1 0 0 0 0 | + | 35 21 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0072 | + | ID FL3FAACS0072 |
| − | KNApSAcK_ID C00014040 | + | KNApSAcK_ID C00014040 |
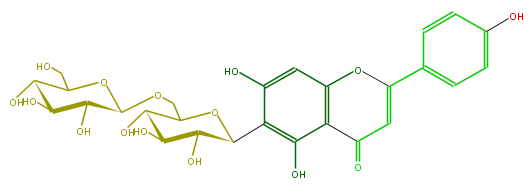
| − | NAME Isovitexin 6''-O-glucoside | + | NAME Isovitexin 6''-O-glucoside |
| − | CAS_RN 191615-66-4 | + | CAS_RN 191615-66-4 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c5)(ccc(c5)C(O1)=CC(c(c2O)c1cc(c(C(O3)C(C(O)C(O)C(COC(C4O)OC(C(C(O)4)O)CO)3)O)2)O)=O)O | + | SMILES c(c5)(ccc(c5)C(O1)=CC(c(c2O)c1cc(c(C(O3)C(C(O)C(O)C(COC(C4O)OC(C(C(O)4)O)CO)3)O)2)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
2.8813 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1669 0.5566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4524 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4524 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1669 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8813 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7379 0.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0234 0.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0234 -0.6809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7379 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6476 0.5315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7379 -1.8329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1669 -1.6655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6830 -0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2704 -1.0982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4721 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6786 -1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0911 -0.4011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8895 -0.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0525 -0.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3981 -0.0297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1662 -0.8667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7191 -0.8391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6435 -1.5290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6631 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3775 0.1829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0920 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0920 1.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3775 1.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6631 1.4204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7072 1.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2240 0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8114 -0.4217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0131 -0.2125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2196 -0.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6321 0.2754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4305 0.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5935 0.4472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9391 0.6468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7072 -0.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2601 -0.1626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1845 -0.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
8 11 1 0 0 0 0
10 12 1 0 0 0 0
5 13 2 0 0 0 0
14 15 1 1 0 0 0
15 16 1 1 0 0 0
17 16 1 1 0 0 0
17 18 1 0 0 0 0
18 19 1 0 0 0 0
19 14 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 0 0 0 0
14 22 1 0 0 0 0
15 23 1 0 0 0 0
16 24 1 0 0 0 0
9 17 1 0 0 0 0
1 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 25 1 0 0 0 0
28 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 21 1 0 0 0 0
S SKP 8
ID FL3FAACS0072
KNApSAcK_ID C00014040
NAME Isovitexin 6''-O-glucoside
CAS_RN 191615-66-4
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c5)(ccc(c5)C(O1)=CC(c(c2O)c1cc(c(C(O3)C(C(O)C(O)C(COC(C4O)OC(C(C(O)4)O)CO)3)O)2)O)=O)O
M END