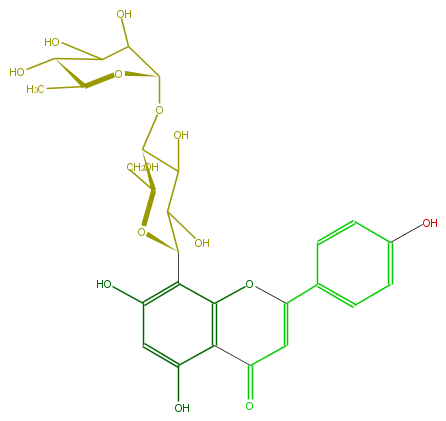
Mol:FL3FAACS0057
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6143 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6143 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6143 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6143 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8999 -3.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8999 -3.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1854 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1854 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1854 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1854 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8999 -1.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8999 -1.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5291 -3.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5291 -3.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2435 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2435 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2435 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2435 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5291 -1.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5291 -1.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5304 -3.9043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5304 -3.9043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3285 -1.4654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3285 -1.4654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6363 -0.4240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6363 -0.4240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9070 -0.8452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9070 -0.8452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1384 -0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1384 -0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9070 0.7794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9070 0.7794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6363 1.2006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6363 1.2006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4050 0.3907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4050 0.3907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2840 2.0113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2840 2.0113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9135 1.5179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9135 1.5179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4976 -0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4976 -0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8782 -1.4917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8782 -1.4917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6051 -1.9114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6051 -1.9114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3321 -1.4917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3321 -1.4917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3321 -0.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3321 -0.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6051 -0.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6051 -0.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8782 -0.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8782 -0.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0585 -0.2330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0585 -0.2330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8999 -3.9400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8999 -3.9400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3940 3.0391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3940 3.0391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7871 2.4702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7871 2.4702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0904 2.7427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0904 2.7427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2980 2.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2980 2.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9113 3.2610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9113 3.2610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4280 3.0131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4280 3.0131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0585 2.7772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0585 2.7772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6532 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6532 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0644 3.9400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0644 3.9400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3543 3.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3543 3.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9728 0.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9728 0.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3307 1.9938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3307 1.9938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 14 13 1 1 0 0 0 | + | 14 13 1 1 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 13 1 1 0 0 0 | + | 18 13 1 1 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 14 1 0 0 0 0 | + | 6 14 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 9 22 1 0 0 0 0 | + | 9 22 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 33 19 1 0 0 0 0 | + | 33 19 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 18 40 1 0 0 0 0 | + | 18 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 45 0.5678 -0.4909 | + | M SBV 1 45 0.5678 -0.4909 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAACS0057 | + | ID FL3FAACS0057 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES c(c3O)c(c(C(O5)C(O)C(O)C(C5CO)OC(O4)C(O)C(O)C(O)C(C)4)c(c32)OC(=CC2=O)c(c1)ccc(O)c1)O | + | SMILES c(c3O)c(c(C(O5)C(O)C(O)C(C5CO)OC(O4)C(O)C(O)C(O)C(C)4)c(c32)OC(=CC2=O)c(c1)ccc(O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.6143 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6143 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8999 -3.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1854 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1854 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8999 -1.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5291 -3.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2435 -2.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2435 -1.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5291 -1.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5304 -3.9043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3285 -1.4654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6363 -0.4240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9070 -0.8452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1384 -0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9070 0.7794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6363 1.2006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4050 0.3907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2840 2.0113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9135 1.5179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4976 -0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8782 -1.4917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6051 -1.9114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3321 -1.4917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3321 -0.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6051 -0.2327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8782 -0.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0585 -0.2330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8999 -3.9400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3940 3.0391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7871 2.4702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0904 2.7427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2980 2.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9113 3.2610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4280 3.0131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0585 2.7772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6532 2.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0644 3.9400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3543 3.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9728 0.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3307 1.9938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
14 13 1 1 0 0 0
14 15 1 0 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 1 1 0 0 0
18 13 1 1 0 0 0
17 19 1 0 0 0 0
16 20 1 0 0 0 0
15 21 1 0 0 0 0
6 14 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
9 22 1 0 0 0 0
25 28 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
34 38 1 0 0 0 0
35 39 1 0 0 0 0
33 19 1 0 0 0 0
40 41 1 0 0 0 0
18 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^ CH2OH
M SBV 1 45 0.5678 -0.4909
S SKP 5
ID FL3FAACS0057
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES c(c3O)c(c(C(O5)C(O)C(O)C(C5CO)OC(O4)C(O)C(O)C(O)C(C)4)c(c32)OC(=CC2=O)c(c1)ccc(O)c1)O
M END