Mol:FL3FAACS0032
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 44 0 0 0 0 0 0 0 0999 V2000 | + | 40 44 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.8180 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8180 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8180 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8180 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2617 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2617 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2946 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2946 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2946 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2946 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2617 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2617 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8509 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8509 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4072 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4072 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4072 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4072 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8509 -0.7513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8509 -0.7513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8509 -2.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8509 -2.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3741 -0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3741 -0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0369 1.7270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0369 1.7270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6425 1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6425 1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3856 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3856 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3787 -0.0299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3787 -0.0299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0846 0.4333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0846 0.4333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2204 1.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2204 1.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2917 2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2917 2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6773 1.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6773 1.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0210 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0210 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2617 -2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2617 -2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0871 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0871 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6733 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6733 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2595 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2595 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2595 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2595 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6733 0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6733 0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0871 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0871 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8454 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8454 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6948 0.6307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6948 0.6307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6948 1.2583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6948 1.2583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1982 0.3401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1982 0.3401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7473 0.6571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7473 0.6571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2964 0.3401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2964 0.3401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2964 -0.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2964 -0.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7473 -0.6110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7473 -0.6110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1982 -0.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1982 -0.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8454 -0.6109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8454 -0.6109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4755 1.4130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4755 1.4130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1899 1.0005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1899 1.0005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 9 23 1 0 0 0 0 | + | 9 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 30 21 1 0 0 0 0 | + | 30 21 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 18 39 1 0 0 0 0 | + | 18 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 43 -5.1711 5.9293 | + | M SBV 1 43 -5.1711 5.9293 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0032 | + | ID FL3FAACS0032 |
| − | KNApSAcK_ID C00006258 | + | KNApSAcK_ID C00006258 |
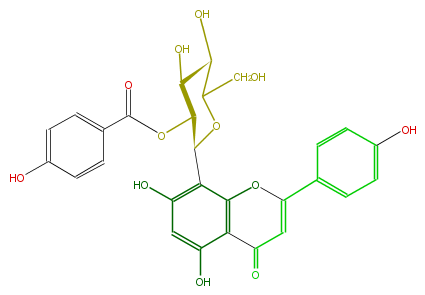
| − | NAME Vitexin 2''-p-hydroxybenzoate | + | NAME Vitexin 2''-p-hydroxybenzoate |
| − | CAS_RN 10576-90-6 | + | CAS_RN 10576-90-6 |
| − | FORMULA C28H24O12 | + | FORMULA C28H24O12 |
| − | EXACTMASS 552.126776232 | + | EXACTMASS 552.126776232 |
| − | AVERAGEMASS 552.48296 | + | AVERAGEMASS 552.48296 |
| − | SMILES OCC(O1)C(O)C(C(OC(c(c5)ccc(c5)O)=O)C1c(c(O)4)c(O2)c(c(c4)O)C(=O)C=C2c(c3)ccc(c3)O)O | + | SMILES OCC(O1)C(O)C(C(OC(c(c5)ccc(c5)O)=O)C1c(c(O)4)c(O2)c(c(c4)O)C(=O)C=C2c(c3)ccc(c3)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 44 0 0 0 0 0 0 0 0999 V2000
-0.8180 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8180 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2617 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2946 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2946 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2617 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8509 -2.0360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4072 -1.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4072 -1.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8509 -0.7513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8509 -2.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3741 -0.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0369 1.7270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6425 1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3856 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3787 -0.0299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0846 0.4333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2204 1.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2917 2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6773 1.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0210 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2617 -2.6781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0871 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6733 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2595 -0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2595 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6733 0.3671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0871 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8454 0.3669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6948 0.6307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6948 1.2583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1982 0.3401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7473 0.6571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2964 0.3401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2964 -0.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7473 -0.6110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1982 -0.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8454 -0.6109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4755 1.4130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1899 1.0005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
9 23 1 0 0 0 0
26 29 1 0 0 0 0
30 21 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 32 1 0 0 0 0
35 38 1 0 0 0 0
18 39 1 0 0 0 0
39 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 CH2OH
M SBV 1 43 -5.1711 5.9293
S SKP 8
ID FL3FAACS0032
KNApSAcK_ID C00006258
NAME Vitexin 2''-p-hydroxybenzoate
CAS_RN 10576-90-6
FORMULA C28H24O12
EXACTMASS 552.126776232
AVERAGEMASS 552.48296
SMILES OCC(O1)C(O)C(C(OC(c(c5)ccc(c5)O)=O)C1c(c(O)4)c(O2)c(c(c4)O)C(=O)C=C2c(c3)ccc(c3)O)O
M END