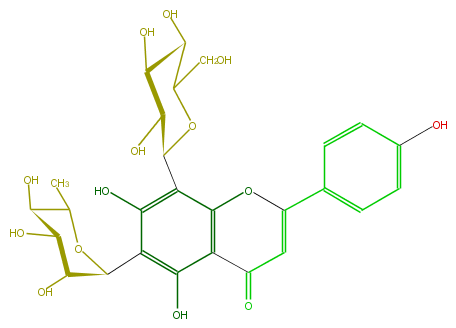
Mol:FL3FAACS0031
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7506 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7506 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7506 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7506 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0361 -2.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0361 -2.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3217 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3217 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3217 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3217 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0361 -0.6349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0361 -0.6349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3928 -2.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3928 -2.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1073 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1073 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1073 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1073 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3928 -0.6349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3928 -0.6349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3928 -2.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3928 -2.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4648 -0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4648 -0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9010 -0.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9010 -0.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6538 -1.0432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6538 -1.0432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4067 -0.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4067 -0.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4067 0.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4067 0.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6538 0.6955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6538 0.6955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9010 0.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9010 0.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1590 0.6952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1590 0.6952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8712 2.3210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8712 2.3210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6489 1.7319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6489 1.7319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3190 0.8833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3190 0.8833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3101 0.0647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3101 0.0647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7151 0.6596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7151 0.6596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1070 1.4019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1070 1.4019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2298 2.9165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2298 2.9165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6923 2.5459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6923 2.5459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8863 0.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8863 0.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0361 -3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0361 -3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2178 -2.3222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2178 -2.3222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4389 -2.3222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4389 -2.3222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9987 -1.7807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9987 -1.7807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1901 -1.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1901 -1.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9690 -1.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9690 -1.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4093 -1.5626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4093 -1.5626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0137 -0.4140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0137 -0.4140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4948 -0.4772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4948 -0.4772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1590 -1.4791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1590 -1.4791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7367 -2.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7367 -2.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5139 1.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5139 1.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1283 3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1283 3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 31 2 1 0 0 0 0 | + | 31 2 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 45 -0.5931 -0.5954 | + | M SBV 1 45 -0.5931 -0.5954 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAACS0031 | + | ID FL3FAACS0031 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES C(C1O)(OC(c(c(O)4)c(c2c(O)c4C(C5O)OC(C(C5O)O)C)OC(c(c3)ccc(O)c3)=CC(=O)2)C(C1O)O)CO | + | SMILES C(C1O)(OC(c(c(O)4)c(c2c(O)c4C(C5O)OC(C(C5O)O)C)OC(c(c3)ccc(O)c3)=CC(=O)2)C(C1O)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.7506 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7506 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0361 -2.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3217 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3217 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0361 -0.6349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3928 -2.2849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1073 -1.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1073 -1.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3928 -0.6349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3928 -2.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4648 -0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9010 -0.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6538 -1.0432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4067 -0.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4067 0.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6538 0.6955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9010 0.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1590 0.6952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8712 2.3210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6489 1.7319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3190 0.8833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3101 0.0647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7151 0.6596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1070 1.4019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2298 2.9165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6923 2.5459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8863 0.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0361 -3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2178 -2.3222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4389 -2.3222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9987 -1.7807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1901 -1.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9690 -1.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4093 -1.5626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0137 -0.4140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4948 -0.4772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1590 -1.4791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7367 -2.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5139 1.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1283 3.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
3 29 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
35 38 1 0 0 0 0
30 39 1 0 0 0 0
31 2 1 0 0 0 0
40 41 1 0 0 0 0
25 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 CH2OH
M SBV 1 45 -0.5931 -0.5954
S SKP 5
ID FL3FAACS0031
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES C(C1O)(OC(c(c(O)4)c(c2c(O)c4C(C5O)OC(C(C5O)O)C)OC(c(c3)ccc(O)c3)=CC(=O)2)C(C1O)O)CO
M END