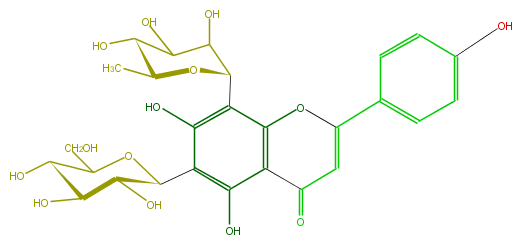
Mol:FL3FAACS0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3449 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3449 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3449 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3449 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6305 -1.3490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6305 -1.3490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0840 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0840 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0840 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0840 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6305 0.3010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6305 0.3010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7984 -1.3490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7984 -1.3490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5128 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5128 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5128 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5128 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7984 0.3010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7984 0.3010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7984 -1.9921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7984 -1.9921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6305 -2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6305 -2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3860 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3860 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1388 -0.0014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1388 -0.0014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8916 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8916 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8916 1.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8916 1.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1388 1.7372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1388 1.7372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3860 1.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3860 1.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8055 1.9281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8055 1.9281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2322 -0.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2322 -0.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5699 -1.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5699 -1.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9078 -1.1410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9078 -1.1410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0073 -1.2470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0073 -1.2470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6430 -0.6643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6430 -0.6643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3580 -1.0086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3580 -1.0086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8055 -1.0744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8055 -1.0744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3317 -1.6044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3317 -1.6044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0799 0.3129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0799 0.3129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1997 -1.6450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1997 -1.6450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5298 1.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5298 1.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1325 0.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1325 0.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3357 1.0521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3357 1.0521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6150 0.9330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6150 0.9330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0455 1.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0455 1.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7585 1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7585 1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1440 1.5370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1440 1.5370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8655 1.0843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8655 1.0843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0389 2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0389 2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3030 2.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3030 2.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8512 -0.5139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8512 -0.5139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6005 0.2352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6005 0.2352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 6 33 1 0 0 0 0 | + | 6 33 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 0.4932 -0.4947 | + | M SBV 1 45 0.4932 -0.4947 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAACS0026 | + | ID FL3FAACS0026 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES C(C(C5O)OC(C(C5O)O)c(c2O)c(O)c(c(O3)c2C(=O)C=C3c(c4)ccc(O)c4)C(O1)C(C(C(O)C1C)O)O)O | + | SMILES C(C(C5O)OC(C(C5O)O)c(c2O)c(O)c(c(O3)c2C(=O)C=C3c(c4)ccc(O)c4)C(O1)C(C(C(O)C1C)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.3449 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3449 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6305 -1.3490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0840 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0840 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6305 0.3010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7984 -1.3490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5128 -0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5128 -0.1115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7984 0.3010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7984 -1.9921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6305 -2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3860 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1388 -0.0014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8916 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8916 1.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1388 1.7372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3860 1.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8055 1.9281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2322 -0.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5699 -1.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9078 -1.1410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0073 -1.2470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6430 -0.6643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3580 -1.0086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8055 -1.0744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3317 -1.6044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0799 0.3129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1997 -1.6450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5298 1.6683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1325 0.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3357 1.0521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6150 0.9330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0455 1.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7585 1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1440 1.5370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8655 1.0843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0389 2.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3030 2.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8512 -0.5139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6005 0.2352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
2 23 1 0 0 0 0
1 28 1 0 0 0 0
22 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
34 38 1 0 0 0 0
35 39 1 0 0 0 0
6 33 1 0 0 0 0
40 41 1 0 0 0 0
25 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 0.4932 -0.4947
S SKP 5
ID FL3FAACS0026
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES C(C(C5O)OC(C(C5O)O)c(c2O)c(O)c(c(O3)c2C(=O)C=C3c(c4)ccc(O)c4)C(O1)C(C(C(O)C1C)O)O)O
M END