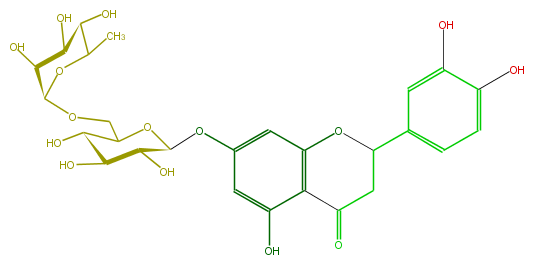
Mol:FL2FACGS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6029 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6029 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6029 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6029 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1119 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1119 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8266 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8266 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8266 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8266 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1119 0.0266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1119 0.0266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5414 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5414 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2562 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2562 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2562 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2562 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5414 0.0266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5414 0.0266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9707 0.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9707 0.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6992 -0.3941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6992 -0.3941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4277 0.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4277 0.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4277 0.8676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4277 0.8676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6992 1.2882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6992 1.2882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9707 0.8676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9707 0.8676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5414 -2.3227 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5414 -2.3227 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1559 1.2881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1559 1.2881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3174 0.0264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3174 0.0264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6992 2.2113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6992 2.2113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1119 -2.4491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1119 -2.4491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7309 -0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7309 -0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2579 -0.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2579 -0.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5767 -0.3805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5767 -0.3805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9195 -0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9195 -0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3971 0.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3971 0.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9930 -0.2103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9930 -0.2103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1778 0.2191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1778 0.2191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2558 -0.2246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2558 -0.2246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9712 -0.6776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9712 -0.6776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0614 -0.8101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0614 -0.8101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6979 1.3306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6979 1.3306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5255 0.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5255 0.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2024 1.2696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2024 1.2696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6179 1.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6179 1.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7902 2.2384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7902 2.2384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1133 1.6566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1133 1.6566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9438 0.3101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9438 0.3101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2589 2.4491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2589 2.4491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1730 1.9826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1730 1.9826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1947 2.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1947 2.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1559 1.7637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1559 1.7637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 38 28 1 0 0 0 0 | + | 38 28 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 32 42 1 0 0 0 0 | + | 32 42 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FACGS0008 | + | ID FL2FACGS0008 |
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C(Oc(c3)cc(O)c(C(=O)5)c3OC(C5)c(c4)cc(c(O)c4)O)2)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C(Oc(c3)cc(O)c(C(=O)5)c3OC(C5)c(c4)cc(c(O)c4)O)2)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.6029 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6029 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1119 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8266 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8266 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1119 0.0266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5414 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2562 -1.2114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2562 -0.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5414 0.0266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9707 0.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6992 -0.3941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4277 0.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4277 0.8676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6992 1.2882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9707 0.8676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5414 -2.3227 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1559 1.2881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3174 0.0264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6992 2.2113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1119 -2.4491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7309 -0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2579 -0.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5767 -0.3805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9195 -0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3971 0.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9930 -0.2103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1778 0.2191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2558 -0.2246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9712 -0.6776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0614 -0.8101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6979 1.3306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5255 0.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2024 1.2696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6179 1.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7902 2.2384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1133 1.6566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9438 0.3101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2589 2.4491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1730 1.9826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1947 2.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1559 1.7637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
1 19 1 0 0 0 0
20 15 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
33 38 1 0 0 0 0
38 28 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
37 41 1 0 0 0 0
32 42 1 0 0 0 0
25 19 1 0 0 0 0
S SKP 5
ID FL2FACGS0008
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C(Oc(c3)cc(O)c(C(=O)5)c3OC(C5)c(c4)cc(c(O)c4)O)2)O)C(C(O)C(O)1)O
M END