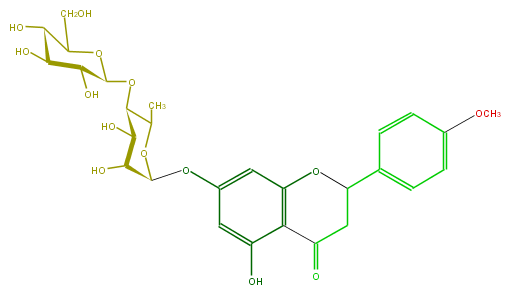
Mol:FL2FABGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1236 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1236 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1236 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1236 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4089 -2.1751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4089 -2.1751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3059 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3059 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3059 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3059 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4089 -0.5245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4089 -0.5245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0207 -2.1751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0207 -2.1751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7354 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7354 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7354 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7354 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0207 -0.5245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0207 -0.5245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4499 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4499 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1784 -0.9452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1784 -0.9452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9068 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9068 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9068 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9068 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1784 0.7371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1784 0.7371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4499 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4499 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0207 -2.8738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0207 -2.8738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8757 -0.5057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8757 -0.5057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4089 -3.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4089 -3.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0476 2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0476 2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9494 1.8749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9494 1.8749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2210 1.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2210 1.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6436 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6436 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8198 2.0957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8198 2.0957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4987 2.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4987 2.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5817 2.6778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5817 2.6778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4449 2.1520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4449 2.1520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0649 1.1783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0649 1.1783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2139 -0.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2139 -0.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6377 -0.7682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6377 -0.7682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8205 -0.1284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8205 -0.1284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6377 0.5153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6377 0.5153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2139 0.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2139 0.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0312 0.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0312 0.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0869 1.4412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0869 1.4412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6377 0.9306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6377 0.9306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5275 0.4695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5275 0.4695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7408 -0.5055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7408 -0.5055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6637 0.7535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6637 0.7535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5817 0.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5817 0.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6097 3.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6097 3.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6918 2.4701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6918 2.4701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 29 1 1 0 0 0 | + | 34 29 1 1 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 29 38 1 0 0 0 0 | + | 29 38 1 0 0 0 0 |
| − | 35 23 1 0 0 0 0 | + | 35 23 1 0 0 0 0 |
| − | 30 18 1 0 0 0 0 | + | 30 18 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 14 39 1 0 0 0 0 | + | 14 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 44 -0.7569 -0.4370 | + | M SBV 1 44 -0.7569 -0.4370 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 46 0.1111 -0.8788 | + | M SBV 2 46 0.1111 -0.8788 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FABGS0006 | + | ID FL2FABGS0006 |
| − | FORMULA C28H34O14 | + | FORMULA C28H34O14 |
| − | EXACTMASS 594.194855796 | + | EXACTMASS 594.194855796 |
| − | AVERAGEMASS 594.56116 | + | AVERAGEMASS 594.56116 |
| − | SMILES C(O)C(C(O)5)OC(C(O)C(O)5)OC(C(O)4)C(C)OC(C(O)4)Oc(c1)cc(O)c(C(=O)2)c1OC(c(c3)ccc(c3)OC)C2 | + | SMILES C(O)C(C(O)5)OC(C(O)C(O)5)OC(C(O)4)C(C)OC(C(O)4)Oc(c1)cc(O)c(C(=O)2)c1OC(c(c3)ccc(c3)OC)C2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.1236 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1236 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4089 -2.1751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3059 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3059 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4089 -0.5245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0207 -2.1751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7354 -1.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7354 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0207 -0.5245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4499 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1784 -0.9452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9068 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9068 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1784 0.7371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4499 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0207 -2.8738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8757 -0.5057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4089 -3.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0476 2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9494 1.8749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2210 1.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6436 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8198 2.0957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4987 2.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5817 2.6778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4449 2.1520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0649 1.1783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2139 -0.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6377 -0.7682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8205 -0.1284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6377 0.5153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2139 0.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0312 0.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0869 1.4412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6377 0.9306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5275 0.4695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7408 -0.5055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6637 0.7535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5817 0.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6097 3.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6918 2.4701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 29 1 1 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
34 37 1 0 0 0 0
29 38 1 0 0 0 0
35 23 1 0 0 0 0
30 18 1 0 0 0 0
39 40 1 0 0 0 0
14 39 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 OCH3
M SBV 1 44 -0.7569 -0.4370
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 ^CH2OH
M SBV 2 46 0.1111 -0.8788
S SKP 5
ID FL2FABGS0006
FORMULA C28H34O14
EXACTMASS 594.194855796
AVERAGEMASS 594.56116
SMILES C(O)C(C(O)5)OC(C(O)C(O)5)OC(C(O)4)C(C)OC(C(O)4)Oc(c1)cc(O)c(C(=O)2)c1OC(c(c3)ccc(c3)OC)C2
M END