Mol:FL2FAAGS0025
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3968 -0.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3968 -0.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1095 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1095 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4000 -0.9958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4000 -0.9958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1168 -1.4052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1168 -1.4052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8294 -0.9895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8294 -0.9895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8258 -0.1645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8258 -0.1645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5457 -1.3988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5457 -1.3988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2584 -0.9832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2584 -0.9832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2547 -0.1582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2547 -0.1582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5384 0.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5384 0.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9085 0.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9085 0.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6245 -0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6245 -0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3374 0.2282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3374 0.2282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3342 1.0531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3342 1.0531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6182 1.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6182 1.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9053 1.0476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9053 1.0476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5468 -2.0421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5468 -2.0421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7714 0.1903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7714 0.1903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1168 -2.1298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1168 -2.1298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9530 1.4104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9530 1.4104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9281 0.4764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9281 0.4764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5154 -0.2383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5154 -0.2383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7171 -0.0291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7171 -0.0291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0764 -0.2560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0764 -0.2560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3362 0.4588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3362 0.4588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1346 0.2497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1346 0.2497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3679 -0.6339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3679 -0.6339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0525 0.0718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0525 0.0718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6024 0.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6024 0.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2698 0.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2698 0.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6570 0.9778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6570 0.9778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4194 1.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4194 1.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0230 2.1298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0230 2.1298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2433 1.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2433 1.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6552 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6552 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4791 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4791 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8916 1.4441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8916 1.4441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7166 1.4441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7166 1.4441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1291 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1291 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7166 0.0152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7166 0.0152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8916 0.0152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8916 0.0152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.9530 0.7296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.9530 0.7296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2168 -0.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2168 -0.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8713 -0.7168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8713 -0.7168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8316 -0.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8316 -0.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 28 43 1 0 0 0 0 | + | 28 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FAAGS0025 | + | ID FL2FAAGS0025 |
| − | KNApSAcK_ID C00014319 | + | KNApSAcK_ID C00014319 |
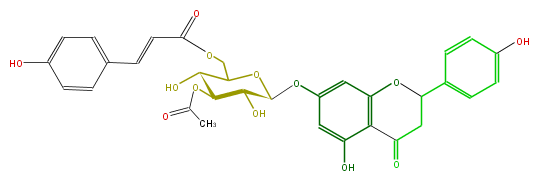
| − | NAME Naringenin 7-[3-acetyl-6-p-coumaroylglucoside];5,7,4'-Trihydroxyflavanone 7-[3-acetyl-6-p-coumaroylglucoside] | + | NAME Naringenin 7-[3-acetyl-6-p-coumaroylglucoside];5,7,4'-Trihydroxyflavanone 7-[3-acetyl-6-p-coumaroylglucoside] |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C32H30O13 | + | FORMULA C32H30O13 |
| − | EXACTMASS 622.168641046 | + | EXACTMASS 622.168641046 |
| − | AVERAGEMASS 622.5728 | + | AVERAGEMASS 622.5728 |
| − | SMILES c(c5)c(ccc(O)5)C=CC(=O)OCC(O1)C(O)C(C(C1Oc(c2)cc(O3)c(C(CC3c(c4)ccc(O)c4)=O)c2O)O)OC(C)=O | + | SMILES c(c5)c(ccc(O)5)C=CC(=O)OCC(O1)C(O)C(C(C1Oc(c2)cc(O3)c(C(CC3c(c4)ccc(O)c4)=O)c2O)O)OC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
1.3968 -0.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1095 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4000 -0.9958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1168 -1.4052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8294 -0.9895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8258 -0.1645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5457 -1.3988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2584 -0.9832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2547 -0.1582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5384 0.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9085 0.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6245 -0.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3374 0.2282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3342 1.0531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6182 1.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9053 1.0476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5468 -2.0421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7714 0.1903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1168 -2.1298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9530 1.4104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9281 0.4764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5154 -0.2383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7171 -0.0291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0764 -0.2560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3362 0.4588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1346 0.2497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3679 -0.6339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0525 0.0718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6024 0.0871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2698 0.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6570 0.9778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4194 1.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0230 2.1298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2433 1.4431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6552 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4791 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8916 1.4441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7166 1.4441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1291 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7166 0.0152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8916 0.0152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.9530 0.7296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2168 -0.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8713 -0.7168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8316 -0.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
14 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
23 27 1 0 0 0 0
22 28 1 0 0 0 0
21 29 1 0 0 0 0
26 30 1 0 0 0 0
30 31 1 0 0 0 0
24 18 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
31 32 1 0 0 0 0
28 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
S SKP 8
ID FL2FAAGS0025
KNApSAcK_ID C00014319
NAME Naringenin 7-[3-acetyl-6-p-coumaroylglucoside];5,7,4'-Trihydroxyflavanone 7-[3-acetyl-6-p-coumaroylglucoside]
CAS_RN -
FORMULA C32H30O13
EXACTMASS 622.168641046
AVERAGEMASS 622.5728
SMILES c(c5)c(ccc(O)5)C=CC(=O)OCC(O1)C(O)C(C(C1Oc(c2)cc(O3)c(C(CC3c(c4)ccc(O)c4)=O)c2O)O)OC(C)=O
M END