Mol:FL2FAAGS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.5801 0.5504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5801 0.5504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2927 0.9660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2927 0.9660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5833 -0.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5833 -0.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3000 -0.6840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3000 -0.6840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0127 -0.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0127 -0.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0090 0.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0090 0.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7290 -0.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7290 -0.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4416 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4416 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4380 0.5630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4380 0.5630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7217 0.9724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7217 0.9724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0917 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0917 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8078 0.5341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8078 0.5341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5207 0.9494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5207 0.9494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5175 1.7744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5175 1.7744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8014 2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8014 2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0885 1.7689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0885 1.7689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7300 -1.3209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7300 -1.3209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0454 0.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0454 0.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3000 -1.4085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3000 -1.4085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1362 2.1316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1362 2.1316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7448 1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7448 1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3322 0.4829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3322 0.4829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5339 0.6921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5339 0.6921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7404 0.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7404 0.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1529 1.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1529 1.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9513 0.9709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9513 0.9709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1847 0.0873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1847 0.0873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8693 0.7930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8693 0.7930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4192 0.8083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4192 0.8083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0865 1.4755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0865 1.4755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4738 1.6990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4738 1.6990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6026 -0.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6026 -0.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1939 -1.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1939 -1.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4265 -0.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4265 -0.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8385 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8385 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6624 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6624 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0749 -0.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0749 -0.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8999 -0.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8999 -0.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3124 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3124 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8999 -2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8999 -2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0749 -2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0749 -2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1362 -1.4696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1362 -1.4696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FAAGS0024 | + | ID FL2FAAGS0024 |
| − | KNApSAcK_ID C00014318 | + | KNApSAcK_ID C00014318 |
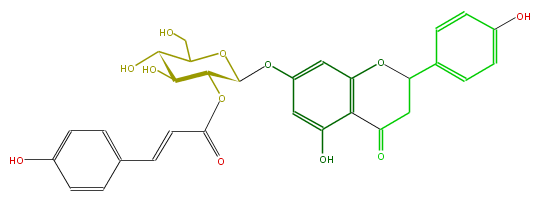
| − | NAME Naringenin 7-(2-p-Coumaroylglucoside);5,7,4'-Trihydroxyflavanone 7-(2-p-Coumaroylglucoside) | + | NAME Naringenin 7-(2-p-Coumaroylglucoside);5,7,4'-Trihydroxyflavanone 7-(2-p-Coumaroylglucoside) |
| − | CAS_RN 172089-70-2 | + | CAS_RN 172089-70-2 |
| − | FORMULA C30H28O12 | + | FORMULA C30H28O12 |
| − | EXACTMASS 580.15807636 | + | EXACTMASS 580.15807636 |
| − | AVERAGEMASS 580.53612 | + | AVERAGEMASS 580.53612 |
| − | SMILES O(C(C4OC(=O)C=Cc(c5)ccc(c5)O)OC(C(C4O)O)CO)c(c3)cc(c(c31)C(=O)CC(c(c2)ccc(O)c2)O1)O | + | SMILES O(C(C4OC(=O)C=Cc(c5)ccc(c5)O)OC(C(C4O)O)CO)c(c3)cc(c(c31)C(=O)CC(c(c2)ccc(O)c2)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
0.5801 0.5504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2927 0.9660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5833 -0.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3000 -0.6840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0127 -0.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0090 0.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7290 -0.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4416 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4380 0.5630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7217 0.9724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0917 0.9439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8078 0.5341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5207 0.9494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5175 1.7744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8014 2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0885 1.7689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7300 -1.3209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0454 0.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3000 -1.4085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1362 2.1316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7448 1.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3322 0.4829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5339 0.6921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7404 0.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1529 1.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9513 0.9709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1847 0.0873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8693 0.7930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4192 0.8083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0865 1.4755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4738 1.6990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6026 -0.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1939 -1.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4265 -0.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8385 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6624 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0749 -0.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8999 -0.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3124 -1.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8999 -2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0749 -2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1362 -1.4696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
14 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
23 27 1 0 0 0 0
22 28 1 0 0 0 0
21 29 1 0 0 0 0
26 30 1 0 0 0 0
30 31 1 0 0 0 0
24 18 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
27 32 1 0 0 0 0
S SKP 8
ID FL2FAAGS0024
KNApSAcK_ID C00014318
NAME Naringenin 7-(2-p-Coumaroylglucoside);5,7,4'-Trihydroxyflavanone 7-(2-p-Coumaroylglucoside)
CAS_RN 172089-70-2
FORMULA C30H28O12
EXACTMASS 580.15807636
AVERAGEMASS 580.53612
SMILES O(C(C4OC(=O)C=Cc(c5)ccc(c5)O)OC(C(C4O)O)CO)c(c3)cc(c(c31)C(=O)CC(c(c2)ccc(O)c2)O1)O
M END