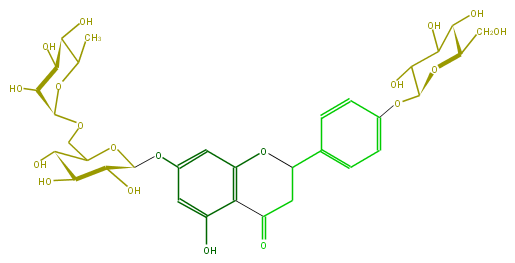
Mol:FL2FAAGS0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 52 57 0 0 0 0 0 0 0 0999 V2000 | + | 52 57 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.4959 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4959 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4959 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4959 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7101 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7101 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9242 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9242 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9242 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9242 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7101 -0.5330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7101 -0.5330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1384 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1384 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6474 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6474 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6474 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6474 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1384 -0.5330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1384 -0.5330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4329 -0.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4329 -0.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2337 -0.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2337 -0.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0346 -0.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0346 -0.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0346 0.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0346 0.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2337 0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2337 0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4329 0.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4329 0.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1384 -3.1159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1384 -3.1159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5406 0.7815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5406 0.7815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0440 -0.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0440 -0.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7101 -3.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7101 -3.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9042 -0.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9042 -0.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3265 -1.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3265 -1.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4944 -1.0040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4944 -1.0040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6917 -0.9953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6917 -0.9953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2750 -0.4118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2750 -0.4118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0029 -0.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0029 -0.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5269 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5269 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4035 -0.8818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4035 -0.8818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0566 -1.3484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0566 -1.3484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8066 -1.5964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8066 -1.5964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3745 1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3745 1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9082 0.4649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9082 0.4649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7803 1.2677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7803 1.2677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2455 1.8860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2455 1.8860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7118 2.5519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7118 2.5519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8397 1.7492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8397 1.7492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1741 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1741 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1288 3.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1288 3.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9954 2.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9954 2.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1391 2.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1391 2.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.8797 1.1958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.8797 1.1958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9335 1.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9335 1.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1470 0.9900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1470 0.9900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5474 1.7110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5474 1.7110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2720 2.1149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2720 2.1149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0586 2.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0586 2.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6580 2.1907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6580 2.1907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4147 1.2986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4147 1.2986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4545 2.9381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4545 2.9381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6311 3.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6311 3.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7938 2.7686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7938 2.7686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8797 1.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8797 1.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 37 27 1 0 0 0 0 | + | 37 27 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 31 41 1 0 0 0 0 | + | 31 41 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 43 18 1 0 0 0 0 | + | 43 18 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 57 -0.5219 -0.6537 | + | M SBV 1 57 -0.5219 -0.6537 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FAAGS0019 | + | ID FL2FAAGS0019 |
| − | FORMULA C33H42O19 | + | FORMULA C33H42O19 |
| − | EXACTMASS 742.2320291619999 | + | EXACTMASS 742.2320291619999 |
| − | AVERAGEMASS 742.67518 | + | AVERAGEMASS 742.67518 |
| − | SMILES C(C(O1)C(O)C(C(O)C(Oc(c2)ccc(C(C6)Oc(c(C6=O)3)cc(OC(C5O)OC(C(O)C5O)COC(C4O)OC(C)C(C(O)4)O)cc3O)c2)1)O)O | + | SMILES C(C(O1)C(O)C(C(O)C(Oc(c2)ccc(C(C6)Oc(c(C6=O)3)cc(OC(C5O)OC(C(O)C5O)COC(C4O)OC(C)C(C(O)4)O)cc3O)c2)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
52 57 0 0 0 0 0 0 0 0999 V2000
-2.4959 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4959 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7101 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9242 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9242 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7101 -0.5330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1384 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6474 -1.8941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6474 -0.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1384 -0.5330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4329 -0.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2337 -0.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0346 -0.5332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0346 0.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2337 0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4329 0.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1384 -3.1159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5406 0.7815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0440 -0.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7101 -3.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9042 -0.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3265 -1.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4944 -1.0040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6917 -0.9953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2750 -0.4118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0029 -0.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5269 -0.3063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.4035 -0.8818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0566 -1.3484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8066 -1.5964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3745 1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9082 0.4649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7803 1.2677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2455 1.8860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7118 2.5519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8397 1.7492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1741 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1288 3.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9954 2.5962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1391 2.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.8797 1.1958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9335 1.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1470 0.9900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5474 1.7110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2720 2.1149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0586 2.9118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6580 2.1907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4147 1.2986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4545 2.9381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6311 3.2547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7938 2.7686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8797 1.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 31 1 1 0 0 0
32 37 1 0 0 0 0
37 27 1 0 0 0 0
35 38 1 0 0 0 0
34 39 1 0 0 0 0
36 40 1 0 0 0 0
31 41 1 0 0 0 0
24 19 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
47 49 1 0 0 0 0
46 50 1 0 0 0 0
43 18 1 0 0 0 0
51 52 1 0 0 0 0
45 51 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 CH2OH
M SBV 1 57 -0.5219 -0.6537
S SKP 5
ID FL2FAAGS0019
FORMULA C33H42O19
EXACTMASS 742.2320291619999
AVERAGEMASS 742.67518
SMILES C(C(O1)C(O)C(C(O)C(Oc(c2)ccc(C(C6)Oc(c(C6=O)3)cc(OC(C5O)OC(C(O)C5O)COC(C4O)OC(C)C(C(O)4)O)cc3O)c2)1)O)O
M END