Mol:FL1DA9NR0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.4950 -1.4353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4950 -1.4353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1181 -1.0756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1181 -1.0756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1181 -0.3562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1181 -0.3562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4950 0.0035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4950 0.0035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8720 -0.3562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8720 -0.3562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8720 -1.0756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8720 -1.0756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1494 -1.4928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1494 -1.4928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4903 -1.1122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4903 -1.1122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1794 -1.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1794 -1.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8392 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8392 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8392 -0.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8392 -0.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5270 0.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5270 0.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2147 -0.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2147 -0.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2147 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2147 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5270 -1.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5270 -1.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1794 -2.1450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1794 -2.1450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5270 -2.0635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5270 -2.0635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3116 0.2038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3116 0.2038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6503 0.8686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6503 0.8686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3872 0.7519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3872 0.7519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3829 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3829 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8525 2.1450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8525 2.1450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5894 2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5894 2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8568 1.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8568 1.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7190 1.2047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7190 1.2047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1181 1.6038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1181 1.6038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0164 0.6895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0164 0.6895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1124 1.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1124 1.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0045 1.2228 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0045 1.2228 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9304 0.6064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9304 0.6064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1385 1.8663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1385 1.8663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5720 0.2950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5720 0.2950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8575 -0.1175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8575 -0.1175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 9 16 2 0 0 0 0 | + | 9 16 2 0 0 0 0 |
| − | 15 17 1 0 0 0 0 | + | 15 17 1 0 0 0 0 |
| − | 11 18 1 0 0 0 0 | + | 11 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 12 1 0 0 0 0 | + | 20 12 1 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 21 28 1 1 0 0 0 | + | 21 28 1 1 0 0 0 |
| − | 19 29 1 1 0 0 0 | + | 19 29 1 1 0 0 0 |
| − | 20 30 1 1 0 0 0 | + | 20 30 1 1 0 0 0 |
| − | 21 31 1 0 0 0 0 | + | 21 31 1 0 0 0 0 |
| − | 13 32 1 0 0 0 0 | + | 13 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 35 -9.7835 5.8165 | + | M SBV 1 35 -9.7835 5.8165 |
| − | S SKP 8 | + | S SKP 8 |
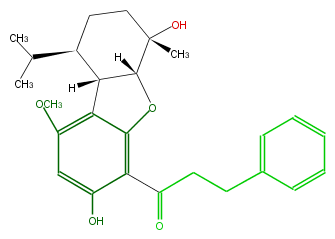
| − | ID FL1DA9NR0011 | + | ID FL1DA9NR0011 |
| − | KNApSAcK_ID C00008115 | + | KNApSAcK_ID C00008115 |
| − | NAME Adunctin E | + | NAME Adunctin E |
| − | CAS_RN 151515-27-4 | + | CAS_RN 151515-27-4 |
| − | FORMULA C26H32O5 | + | FORMULA C26H32O5 |
| − | EXACTMASS 424.224974134 | + | EXACTMASS 424.224974134 |
| − | AVERAGEMASS 424.52927999999997 | + | AVERAGEMASS 424.52927999999997 |
| − | SMILES c(c23)(c(cc(c2C([H])(C4C(C)C)C(C(C)(O)CC4)([H])O3)OC)O)C(CCc(c1)cccc1)=O | + | SMILES c(c23)(c(cc(c2C([H])(C4C(C)C)C(C(C)(O)CC4)([H])O3)OC)O)C(CCc(c1)cccc1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
2.4950 -1.4353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1181 -1.0756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1181 -0.3562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4950 0.0035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8720 -0.3562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8720 -1.0756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1494 -1.4928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4903 -1.1122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1794 -1.4989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8392 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8392 -0.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5270 0.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2147 -0.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2147 -1.1179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5270 -1.5150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1794 -2.1450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5270 -2.0635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3116 0.2038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6503 0.8686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3872 0.7519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3829 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8525 2.1450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5894 2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8568 1.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7190 1.2047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1181 1.6038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0164 0.6895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1124 1.2792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0045 1.2228 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
-1.9304 0.6064 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
0.1385 1.8663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5720 0.2950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8575 -0.1175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
9 16 2 0 0 0 0
15 17 1 0 0 0 0
11 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
20 12 1 0 0 0 0
19 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 20 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 0 0 0 0
25 27 1 0 0 0 0
21 28 1 1 0 0 0
19 29 1 1 0 0 0
20 30 1 1 0 0 0
21 31 1 0 0 0 0
13 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 ^OCH3
M SBV 1 35 -9.7835 5.8165
S SKP 8
ID FL1DA9NR0011
KNApSAcK_ID C00008115
NAME Adunctin E
CAS_RN 151515-27-4
FORMULA C26H32O5
EXACTMASS 424.224974134
AVERAGEMASS 424.52927999999997
SMILES c(c23)(c(cc(c2C([H])(C4C(C)C)C(C(C)(O)CC4)([H])O3)OC)O)C(CCc(c1)cccc1)=O
M END